NOTA: Para descargar los datos, dar click en el boton. Aparece una pantalla con los datos, luego dar click derecho y poner guardar como (save as). Guardar el txt donde van a trabajar
R.1
Llamo los datos, los coloco en un dataframe y convierto las columnas en variables
reg.data<-read.table("tannin.txt",header=T)
attach(reg.data)
names(reg.data)
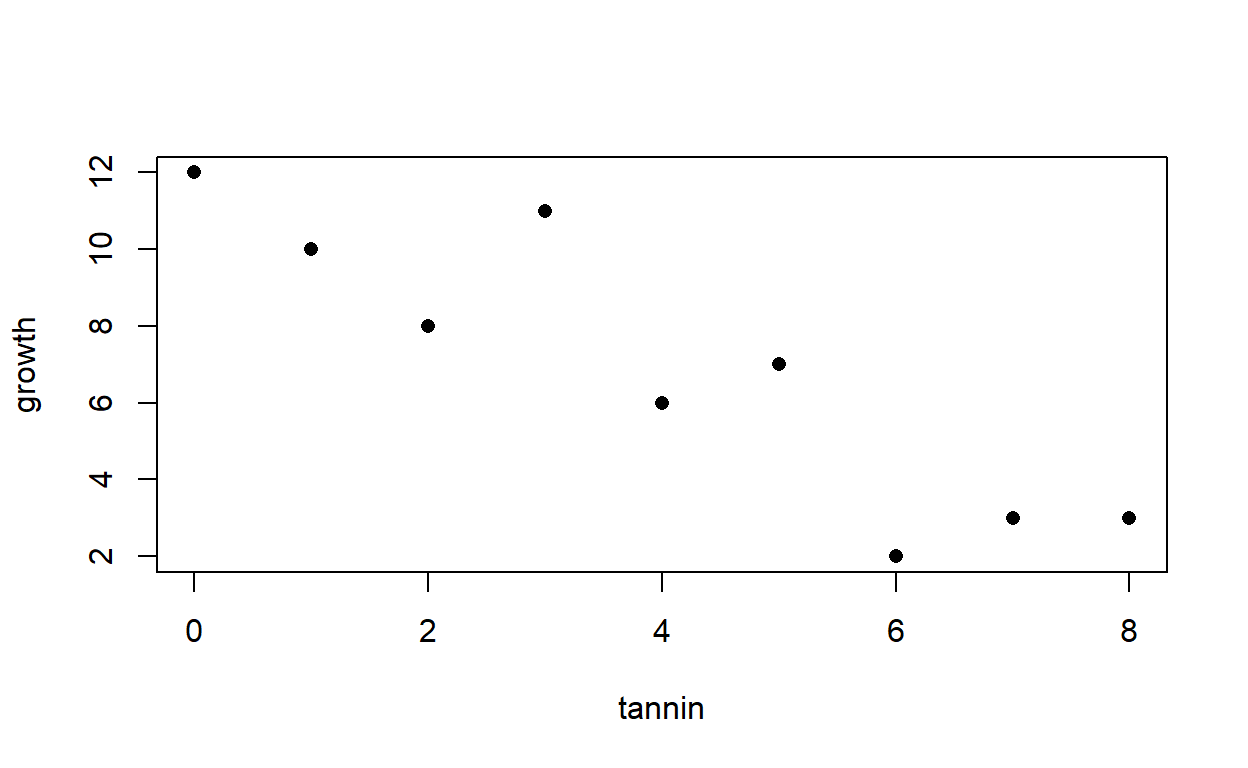
Gráfico
¿La tendencia de la variable de respuesta es a incrementar o a disminuir con la explicatoria? tendencia a disminuir
¿Es factible que los datos sean explicados por una línea horizontal? H0.
La H0 no parece factible, entonces b es probablemente dif de 0 y negativa
¿Si existe una tendencia es recta o curva? relación recta, entonces proponemos el modelo \(y=a+bx+e\)
¿La dispersión de los datos es uniforme a lo largo de la línea o cambia sistemáticamente con la variable explicatoria?. Dispersión muy uniforme, la ordenada al origen es dif de 0 entonces a es prob mayor que 0.
¿A ojo cuales son los valores de a y b? Como podemos hacer este proceso sistemático y preciso?
C.1
R.2
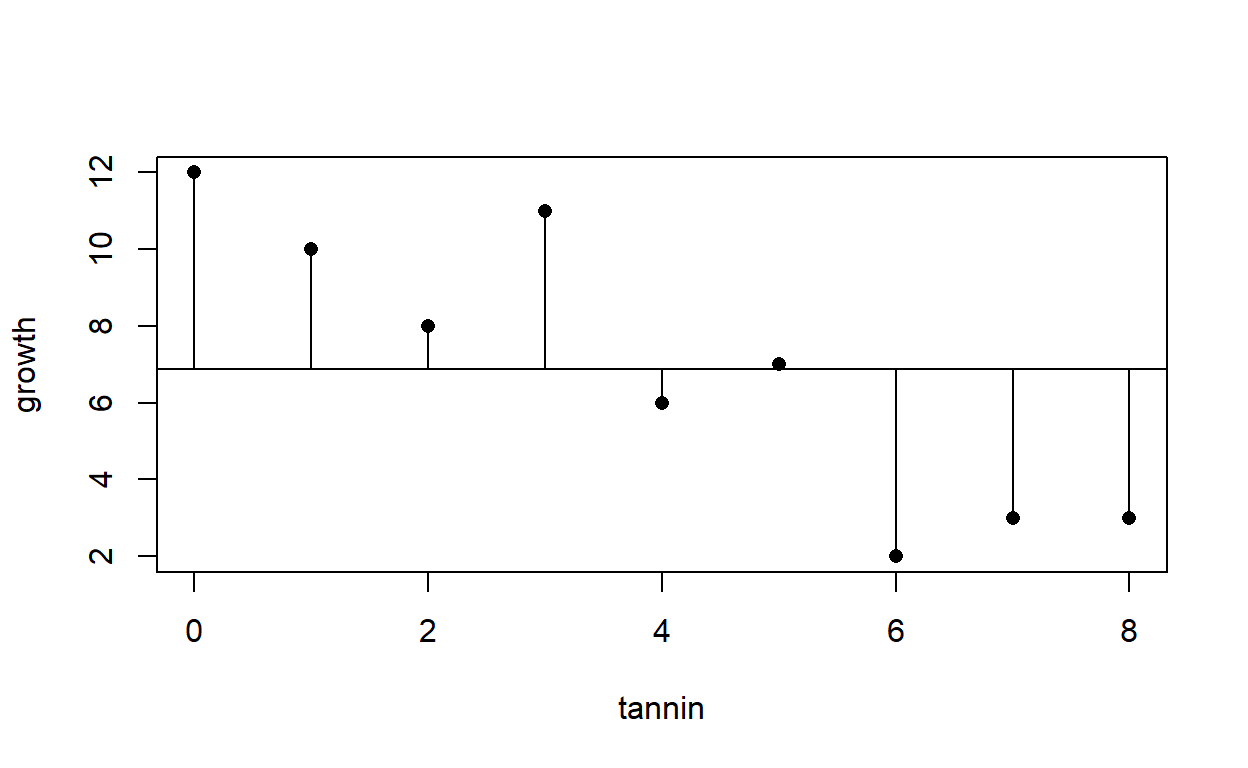
Y la variación total de y es la dispersión de los datos alrededor de y barra.
La Suma de Cuadrados Total es \(SCT= \sum(y-\overline{y})^2\)
plot(tannin,growth,pch=16)
abline(mean(growth),0)
for (i in 1:9) lines(c(tannin[i],tannin[i]),c(growth[i],mean(growth)))
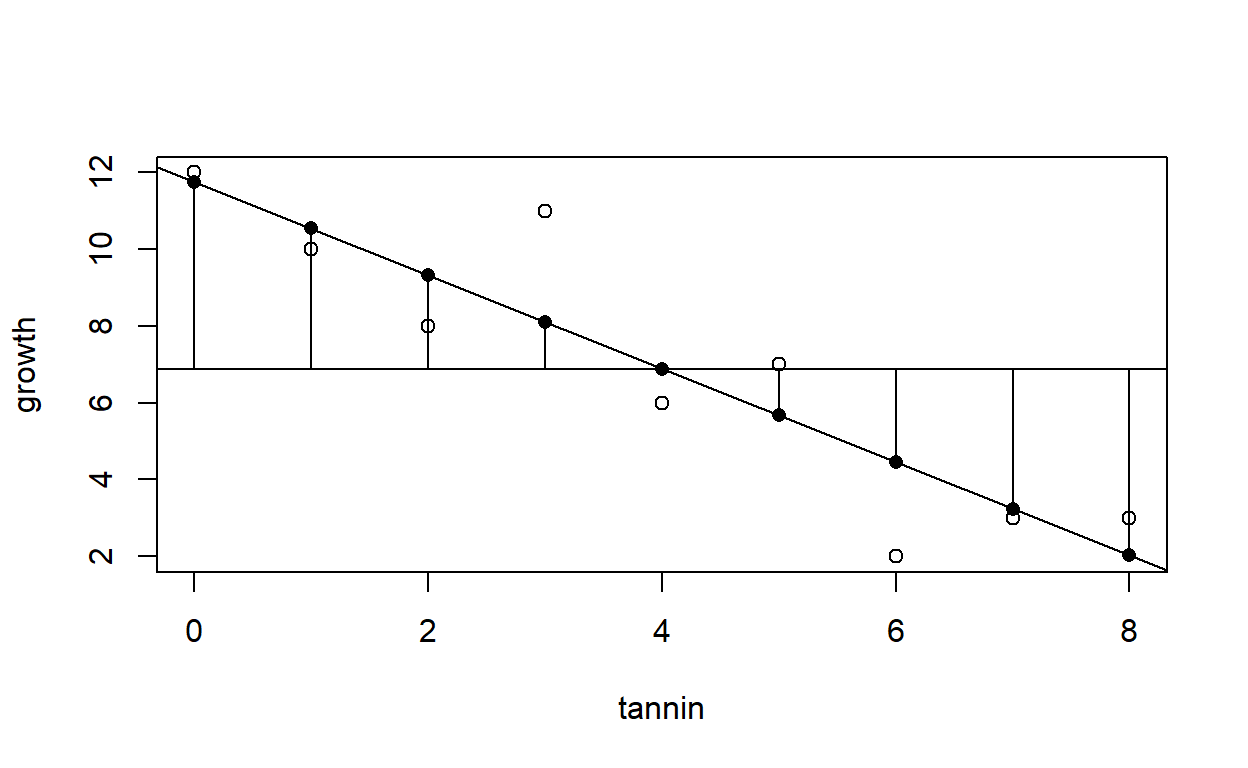
La mejor recta ajustada por el método de mínimos cuadrados es aquella que minimiza la Suma de Cuadrados de las desviaciones de los valores de y de la línea ajustada \(\hat{y}\), \(SCE=\sum(y - \hat{y})^2\)
ysomb <- predict(lm(growth ~ tannin))
plot(tannin,growth,pch=16)
abline(lm(growth~tannin))
for(i in 1:9) lines(c(tannin[i], tannin[i]), c(growth[i], ysomb[i]))

C.2
R.3
Ahora bien, una tercera cantidad es la Suma de Cuadrados de la Regresión (es decir del efecto de la variable predictora)
\[SCR = SCTotal - SCError\]
plot(tannin, growth, type = "n")
abline(mean(growth), 0)
modelito <- lm(growth ~ tannin)
abline(modelito)
for(i in 1:9) lines(c(tannin[i], tannin[i]), c(mean(growth), predict(modelito)[i]))
points(tannin,predict(modelito), pch = 16)
points(tannin, growth)
C.3
R.4
Empezamos a ajustar los modelos: modelo nulo - solo la media
[1] "coefficients" "residuals" "effects" "rank"
[5] "fitted.values" "assign" "qr" "df.residual"
[9] "call" "terms" "model" anova(Nulo)
Analysis of Variance Table
Response: growth
Df Sum Sq Mean Sq F value Pr(>F)
Residuals 8 108.89 13.611 Nulo$df.residual
[1] 8Nulo$coefficients
(Intercept)
6.888889 Nulo$fitted.values
1 2 3 4 5 6 7
6.888889 6.888889 6.888889 6.888889 6.888889 6.888889 6.888889
8 9
6.888889 6.888889 Es decir solo se ha ajustado la media que no ofrece información importante
Agregamos el efecto del tannin
Tanino <- update(Nulo, . ~ . + tannin)
Tanino
Call:
lm(formula = growth ~ tannin)
Coefficients:
(Intercept) tannin
11.756 -1.217 anova(Tanino)
Analysis of Variance Table
Response: growth
Df Sum Sq Mean Sq F value Pr(>F)
tannin 1 88.817 88.817 30.974 0.0008461 ***
Residuals 7 20.072 2.867
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1Tanino$coefficients
(Intercept) tannin
11.755556 -1.216667 summary(Tanino)
Call:
lm(formula = growth ~ tannin)
Residuals:
Min 1Q Median 3Q Max
-2.4556 -0.8889 -0.2389 0.9778 2.8944
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 11.7556 1.0408 11.295 9.54e-06 ***
tannin -1.2167 0.2186 -5.565 0.000846 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.693 on 7 degrees of freedom
Multiple R-squared: 0.8157, Adjusted R-squared: 0.7893
F-statistic: 30.97 on 1 and 7 DF, p-value: 0.0008461O bien pedimos la secuencia de ajustes, que produce estos cambios en devianza
anova(Nulo, Tanino)
Analysis of Variance Table
Model 1: growth ~ 1
Model 2: growth ~ tannin
Res.Df RSS Df Sum of Sq F Pr(>F)
1 8 108.889
2 7 20.072 1 88.817 30.974 0.0008461 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1summary(Tanino)
Call:
lm(formula = growth ~ tannin)
Residuals:
Min 1Q Median 3Q Max
-2.4556 -0.8889 -0.2389 0.9778 2.8944
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 11.7556 1.0408 11.295 9.54e-06 ***
tannin -1.2167 0.2186 -5.565 0.000846 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.693 on 7 degrees of freedom
Multiple R-squared: 0.8157, Adjusted R-squared: 0.7893
F-statistic: 30.97 on 1 and 7 DF, p-value: 0.0008461Si queremos, podemos guardar los valores ajustados y los residuales en la base de datos:
reg.data$ajustados <- fitted.values(Tanino)
reg.data$residuales <- residuals(Tanino)
reg.data
growth tannin ajustados residuales
1 12 0 11.755556 0.2444444
2 10 1 10.538889 -0.5388889
3 8 2 9.322222 -1.3222222
4 11 3 8.105556 2.8944444
5 6 4 6.888889 -0.8888889
6 7 5 5.672222 1.3277778
7 2 6 4.455556 -2.4555556
8 3 7 3.238889 -0.2388889
9 3 8 2.022222 0.9777778Para inspeccionar qué tan bueno es el modelo existen algunos recursos gráficos donde se examinan la distribución de los residuales y los puntos extremos que que pueden “cargar” el valor numérico de los parámetros:
Extra
Una función muy interesante para evaluar un modelo es
check_model del paquete performance(Lüdecke et al. 2021). Recuerda que
puedes instalarlo así:
# Debido a que performance requiere del paquete see para graficar, es bueno instalar ambos
install.packages(c("performance", "see"))
Cargamos las librerias
Ahora usamos la función
check_model(Tanino)
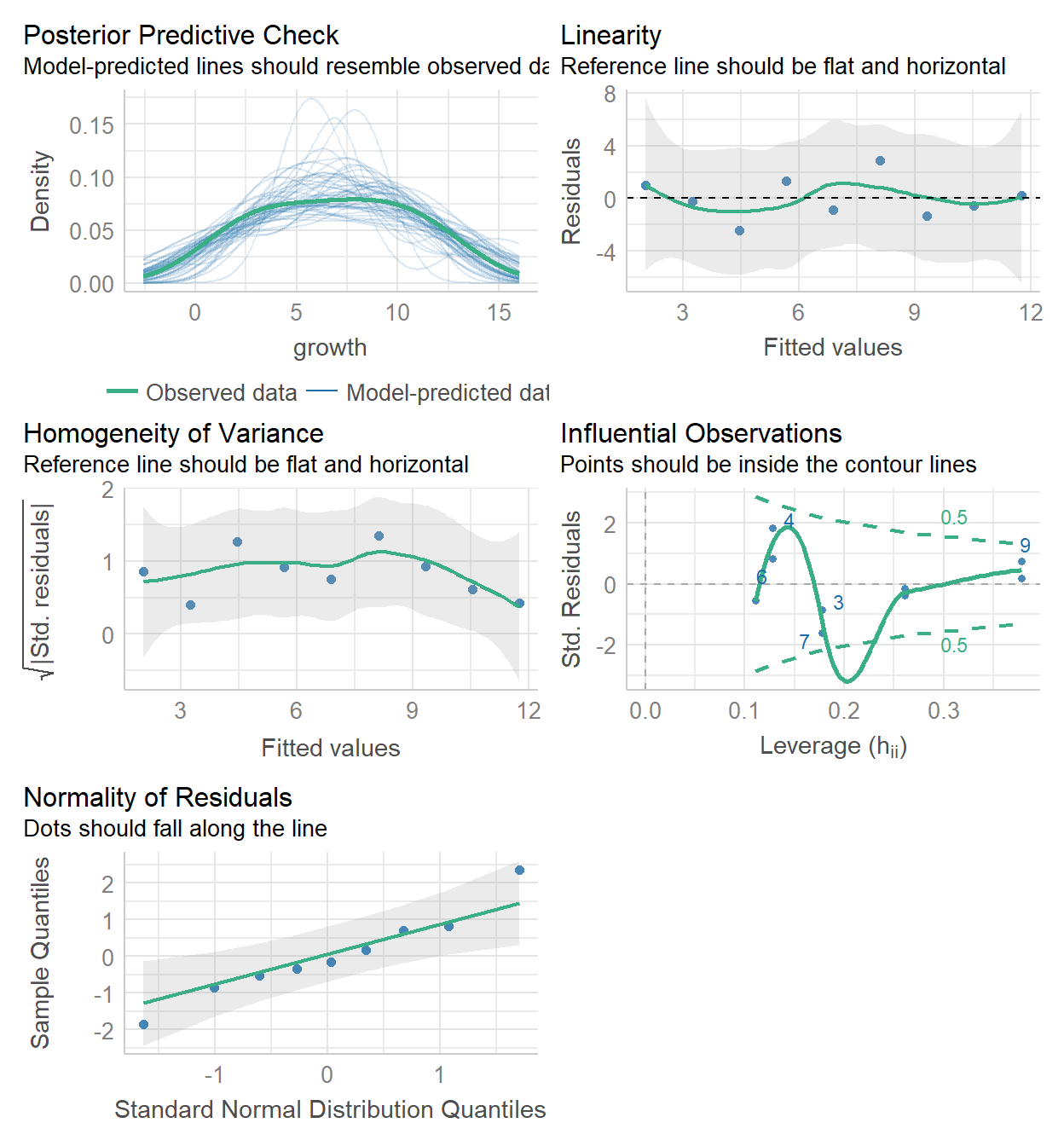
Si son fans de el diagnóstico numérico:
# Por defecto usa los residuales estandarizados
check_normality(Tanino)
OK: residuals appear as normally distributed (p = 0.990).shapiro.test(rstandard(Tanino))
Shapiro-Wilk normality test
data: rstandard(Tanino)
W = 0.98664, p-value = 0.9896check_heteroskedasticity(Tanino) # Breusch-Pagan test (1979).
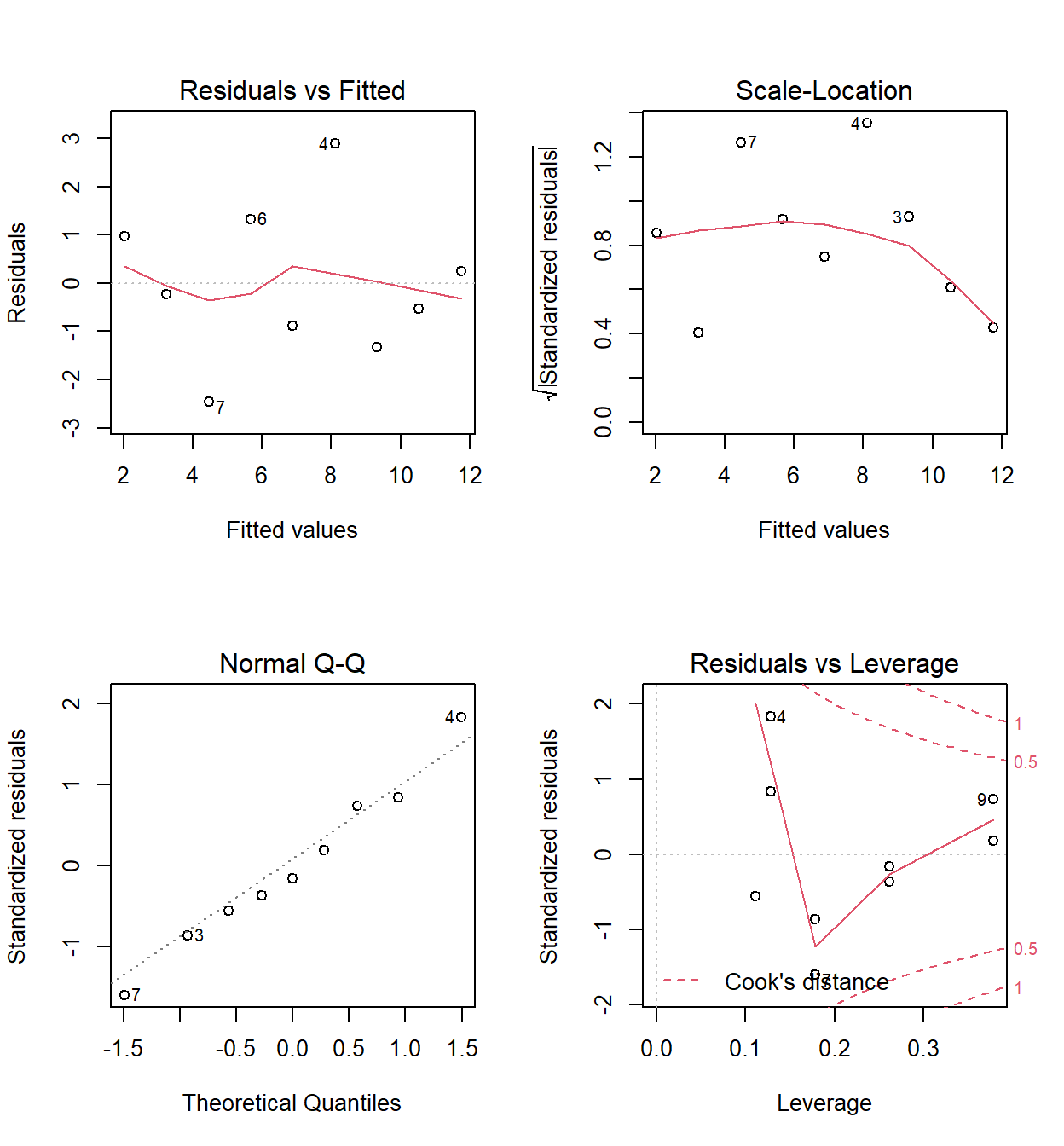
OK: Error variance appears to be homoscedastic (p = 0.843).check_outliers(Tanino)
OK: No outliers detected.
- Based on the following method and threshold: cook (0.77).
- For variable: (Whole model)Examinamos un modelo sin el dato extremo:
Call:
lm(formula = growth[-7] ~ tannin[-7])
Residuals:
Min 1Q Median 3Q Max
-1.4549 -0.9572 -0.1622 0.4572 2.6622
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 11.6892 0.8963 13.042 1.25e-05 ***
tannin[-7] -1.1171 0.1956 -5.712 0.00125 **
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.457 on 6 degrees of freedom
Multiple R-squared: 0.8446, Adjusted R-squared: 0.8188
F-statistic: 32.62 on 1 and 6 DF, p-value: 0.001247No ganamos gran cosa
Extra
Podemos obtener los resultados del modelo lineal en formato de
dataframe con el paquete broom(Robinson,
Hayes, and Couch 2022) y la función tidy
install.packages("broom")
# A tibble: 2 x 5
term estimate std.error statistic p.value
<chr> <dbl> <dbl> <dbl> <dbl>
1 (Intercept) 11.7 0.896 13.0 0.0000125
2 tannin[-7] -1.12 0.196 -5.71 0.00125 De esta forma podemos reportarla de manera más ordenada en un documento Rmarkdown, Quarto o simplemente guardarla como un csv para luego copiarla a mano en un documento word
Así se puede guardar
write.csv(coeftable, # objeto a guardar
file = "modelo2.csv") # nombre del archivo
Para predecir valores usamos:
[1] "coeftable" "i" "modelito" "Nulo" "reg.data"
[6] "Sindat7" "Tanino" "ysomb" Fin