Script multioccu_script.R
1. Instalar y cargar los paquetes —-
Recuerden instalar la versión de desarrollo de camtrapR
remotes::install_github("jniedballa/camtrapR")
Lo primero es cargar todas las librerias necesarias. > Recuerden que para instalar rjags necesitan instalar en su maquina el programa JAGS
library(camtrapR) # Datos de cámaras y modelos
library(rjags) # Para correr el modelo
library(SpadeR) # Riqueza Chao2
library(tidyverse) # Manipular datos
library(nimble) # LEnguaje BUGS
library(nimbleEcology) # Nimble enfocado en jerárquicos
library(bayesplot) # gráficos estimaciones bayesianas
library(SpadeR) # Riqueza Chao2
library(beepr) # Opcional para avisar R termine
library(tictoc) # Opcional para tomar el tiempo de la función
library(extrafont) #opcional para cambiar la fuente
library(snowfall)
2. Cargar los datos —-
Formato de los datos
Vamos a usar los mismos datos que del ejemplo anterior. Seguimos un procedimiento similar para cargar los datos.
# Cargamos la tabla de registros de las especies
registers <- read.csv("Data/Survey/recordTable_OC.csv")
# Cargamos la tabla de operación de cámaras
CToperation <- read.csv("Data/Survey/CTtable_OC.csv")
# Generamos la matríz de operación de las cámaras
camop <- cameraOperation(CTtable= CToperation, # Tabla de operación
stationCol= "Station", # Columna que define la estación
setupCol= "Setup_date", #Columna fecha de colocación
retrievalCol= "Retrieval_date", #Columna fecha de retiro
hasProblems= T, # Hubo fallos de cámaras
dateFormat= "%Y-%m-%d") # Formato de las fechas
# Función para generar las historias de detección para todas las especies seleccionadas
DetHist_list <- lapply(unique(registers$Species), FUN = function(x) {
detectionHistory(
recordTable = registers, # Tabla de registros
camOp = camop, # Matriz de operación de cámaras
stationCol = "Station",
speciesCol = "Species",
recordDateTimeCol = "DateTimeOriginal",
recordDateTimeFormat = "%d/%m/%Y",
species = x, # la función reemplaza x por cada una de las especies
occasionLength = 10, # Colapso de las historias a 10 ías
day1 = "station", #inicie en la fecha de cada estación
datesAsOccasionNames = FALSE,
includeEffort = TRUE,
scaleEffort = TRUE,
timeZone = "America/Mexico_City"
)}
)
# Se genera una lista con cada historia de detección y el esfuerzo de muestreo, ahora le colocaremos los nombres para saber a cual especie corresponde
names(DetHist_list) <- unique(registers$Species)
# Finalmente creamos una lista nueva donde estén solo las historias de detección
ylist <- lapply(DetHist_list, FUN = function(x) x$detection_history)
Terminaremos con el objeto ylist que cotiente todas las historias de detección
covariables
## Covariables
#Cargamos la base de covariables
covars <- read.csv("Data/Covs/stdcovs_OC.csv")
identical(nrow(ylist[[1]]), nrow(covars)) # Verificar que tengan el mismo número de filas
[1] TRUEFinalmente unimos todo en un objeto lista con los datos que requiere el modelo
# Generamos la base de datos para el modelo
data_list <- list(ylist = ylist, # Historias de detección
siteCovs = covars, # Covariables de sitio
obsCovs = list(effort = DetHist_list[[1]]$effort)) # agregamos el esfuerzo de muestreo como covariable de observación
str(data_list)
List of 3
$ ylist :List of 17
..$ Conepatus leuconotus : num [1:67, 1:19] 0 NA 0 0 1 0 0 0 0 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Lynx rufus : num [1:67, 1:19] 0 NA 0 0 0 0 0 0 1 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Sylvilagus floridanus : num [1:67, 1:19] 1 NA 0 0 1 0 0 0 1 1 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Urocyon cinereoargenteus: num [1:67, 1:19] 1 NA 0 0 1 0 0 0 1 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Canis latrans : num [1:67, 1:19] 0 NA 0 0 0 0 0 0 1 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Mephitis macroura : num [1:67, 1:19] 0 NA 0 0 0 0 0 0 0 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Odocoileus virginianus : num [1:67, 1:19] 0 NA 1 0 1 0 0 0 0 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Canis lupus familiaris : num [1:67, 1:19] 0 NA 0 0 0 0 0 0 0 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Pecari tajacu : num [1:67, 1:19] 0 NA 0 0 0 0 0 0 0 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Roedores : num [1:67, 1:19] 0 NA 0 0 1 0 0 0 0 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Dasypus novemcinctus : num [1:67, 1:19] 0 NA 0 0 0 0 0 0 0 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Spilogale angustigrons : num [1:67, 1:19] 0 NA 0 0 0 0 0 0 0 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Bassariscus astutus : num [1:67, 1:19] 0 NA 0 0 0 0 0 0 0 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Procyon lotor : num [1:67, 1:19] 0 NA 0 0 0 0 0 0 0 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Capra hircus : num [1:67, 1:19] 0 NA 0 0 0 0 0 0 0 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Puma yagouaroundi : num [1:67, 1:19] 0 NA 0 0 0 0 0 0 0 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
..$ Nasua narica : num [1:67, 1:19] 0 NA 0 0 0 0 0 0 0 0 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...
$ siteCovs:'data.frame': 67 obs. of 11 variables:
..$ X : int [1:67] 1 2 3 4 5 6 7 8 9 10 ...
..$ Station : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
..$ Cam : chr [1:67] "MoultrieA30" "Moultrie" "MoultrieA30" "Primos" ...
..$ ...1 : num [1:67] -1.69 -1.64 -1.59 -1.54 -1.49 ...
..$ Vertcover_50: num [1:67] -0.474 0.652 -0.924 -0.924 -1.151 ...
..$ Dcrops : num [1:67] 0.677 1.462 2.219 0.46 1.172 ...
..$ MSAVI : num [1:67] 0.732 -0.867 -0.584 -0.143 0.213 ...
..$ Slope : num [1:67] -0.2196 -0.9927 -1.0259 1.4184 -0.0597 ...
..$ Cluster : num [1:67] -1.06 -1.06 -1.06 -1.06 -1.06 ...
..$ Effort : num [1:67] -0.2081 -1.4605 -0.0719 -0.8888 -0.0719 ...
..$ Dpop_G : num [1:67] -0.4261 -0.3167 -0.1805 -0.1923 -0.0948 ...
$ obsCovs :List of 1
..$ effort: num [1:67, 1:19] 0.0626 NA 0.0626 0.0626 0.0626 ...
.. ..- attr(*, "dimnames")=List of 2
.. .. ..$ : chr [1:67] "C1T2P1" "C1T2P11" "C1T2P21" "C1T4P1" ...
.. .. ..$ : chr [1:19] "o1" "o2" "o3" "o4" ...3. Modelo multi-especie —-
Creando el modelo
CamtrapR permite ajustar modelos multi-especie en JAGS y Nimble (es decir en lenguaje BUGS), nosotros vamos a usar JAGS ya que la versión de Nimble aun no permite estimar parámetro N de riqueza de especies
# Usaremos la función ` communityModel`
# Generemos el modelo
comu_model <- communityModel(data_list, # la lista de datos
occuCovs = list(ranef = "Dcrops"), # La covariables de sitio
detCovsObservation = list(fixed = "effort"), #Covariables de observación
intercepts = list(det = "ranef", occu = "ranef"),
augmentation = c(full = 30),# Número aumentado de especies
modelFile = "multmod")# Guardamos la especificación en un archivo
summary(comu_model)
commOccu object (for JAGS)
30 species, 67 stations, 19 occasions
758 occasions with effort
Number of detections (by species): 0 - 146
Available site covariates:
X, Station, Cam, ...1, Vertcover_50, Dcrops, MSAVI, Slope, Cluster, Effort, Dpop_G
Used site covariates:
Dcrops
Available site-occasion covariates:
effort Hora de correr el modelo. En este caso no corran la función porque dura alrededor de 56 min
fit.commu <- fit(comu_model,
n.iter = 22000,
n.burnin = 2000,
thin = 2,
chains = 3,
cores = 3,
quiet = T
);beep(sound = 4)
# Duración 56 min aprox
3.1 Resultados del modelo
modresult <- summary(fit.commu)[["statistics"]]
Veamos el resultado gráfico
Otra gran ventaja de CamtrapR es que permite gráficar de manera muy sencilla la predicción posterior del modelo. Veamos que pasa con la ocupación de cada especie
plot_effects(comu_model, # El modelo
fit.commu, # El objeto ajustado
submodel = "state") # el parámetro de interés
$Dcrops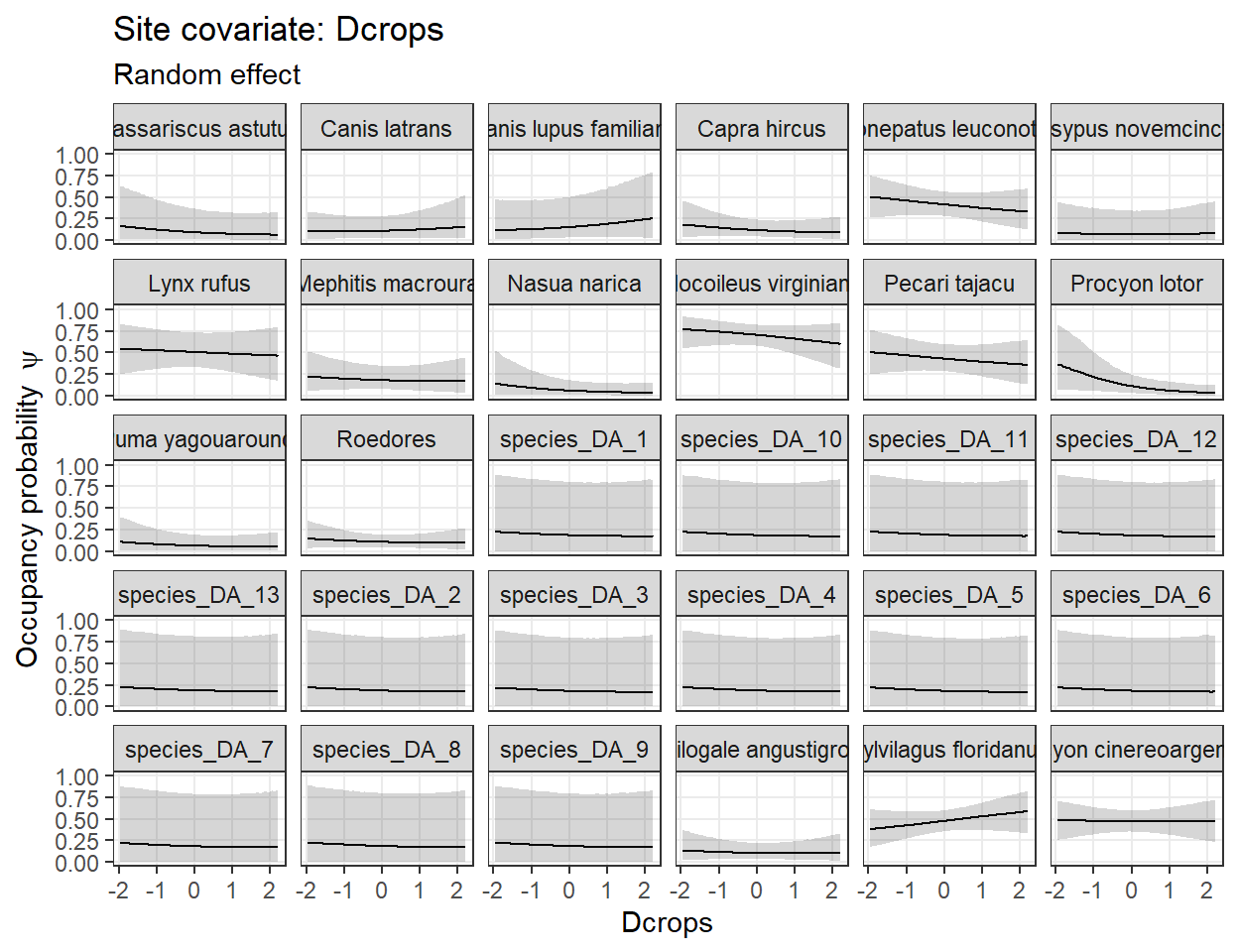
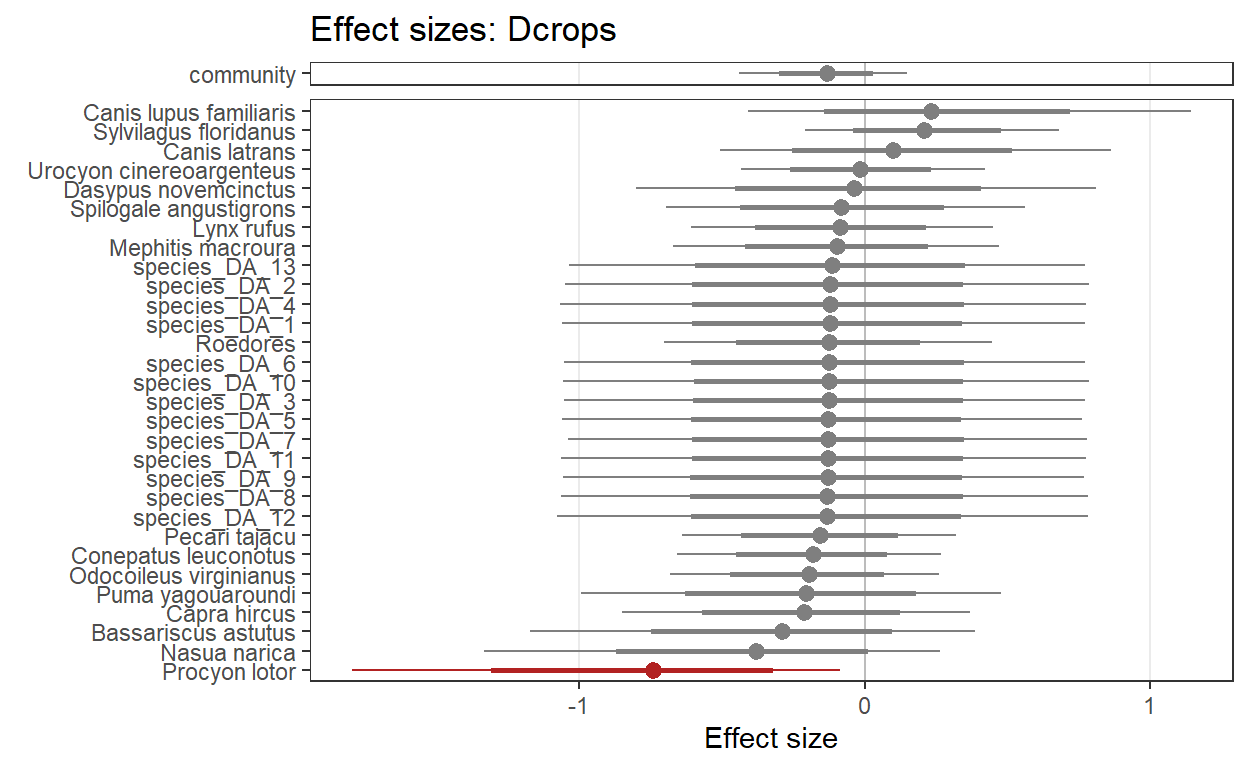
Ahora los coeficientes
Realizamos el mismo procedimiento para el submodelo de detección
y sus respectivos coeficientes3.2 La riqueza de especies
Lo que vinimos a buscar fue la riqueza de especies. La riqueza estimada de especies es ~20 sps
(riqueza_est <- modresult["Ntotal",])
Mean SD Naive SE Time-series SE
19.96107056 2.95338837 0.01705054 0.08235277 # Veamos el gráfico de la distribución posterior
mcmc_areas(fit.commu, # objeto jags
pars= "Ntotal", # parámetro de interés
point_est = "mean",
prob = 0.95) # intervalos de credibilidad
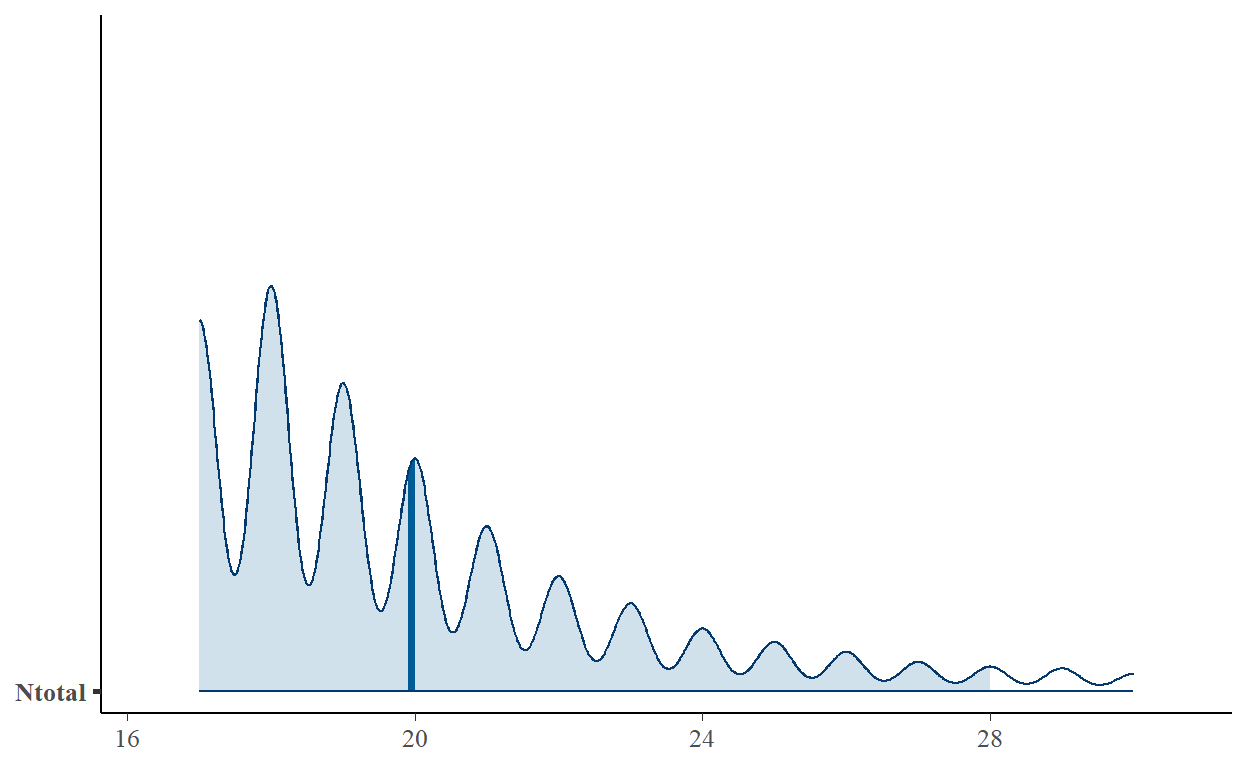
La estimación no se ve muy bien, hay que verificar los trace plots.
Debería verse como un cesped, muy probablemente necesitamos muchas mas iteraciones para este modelo.
mcmc_trace(fit.commu, pars = "Ntotal")
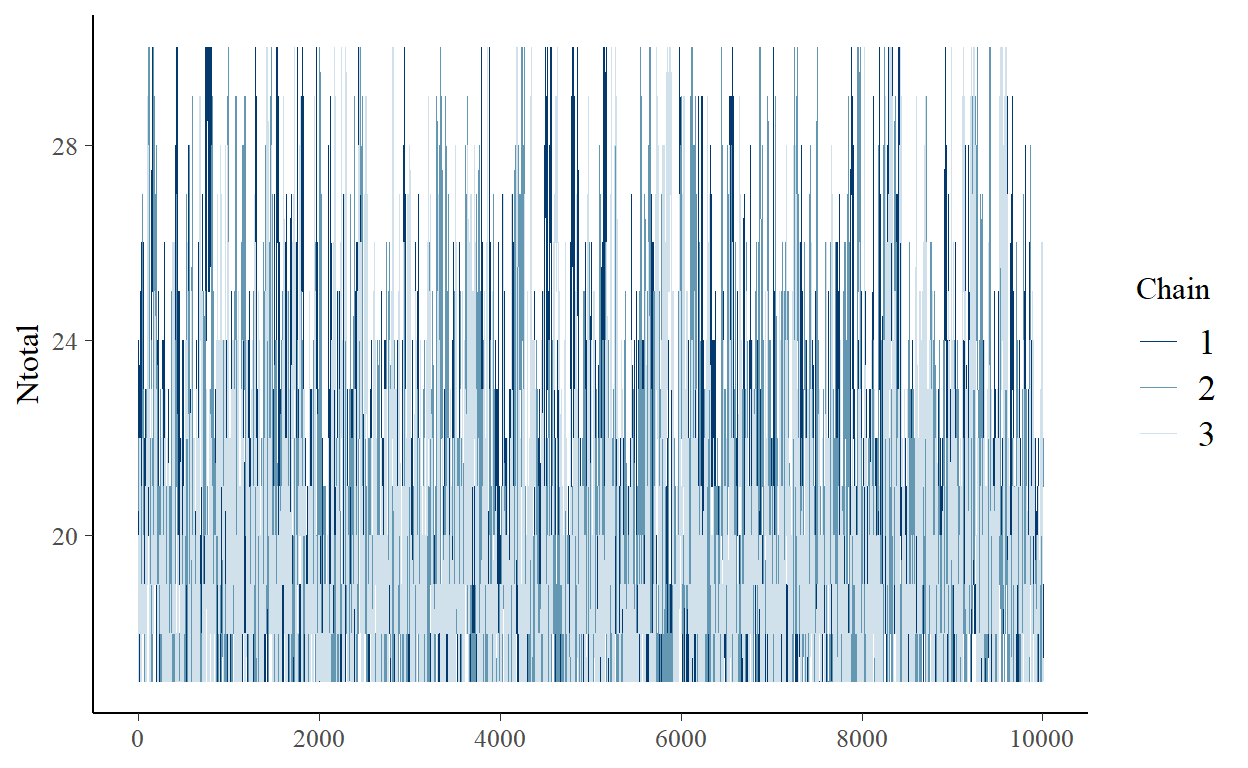
gd <- as.data.frame(gelman.diag(fit.commu, multivariate = FALSE)[[1]])
gd["Ntotal",]
Point est. Upper C.I.
Ntotal 1.001335 1.002693La prueba de Gelman-Rubin debe ser ~1 para considerar que hay buena convergencia. Aunque tenemos un valor bueno para Ntotal, hay varios valores de omega con NA, eso puede estar causando los problemas.
Ajustamos nuestro primer modelo de multi-especie.
Recordemos que N es la riqueza estimada a un área mayor de nuestro muestreo (área que no conocemos)
N depende de si cumplimos los supuestos del área - El muestreo es aleatorio. El área de muestreo debe representar la región - En caso contrario N representa el número de especies un área hipotética con las mismas condiciones - Si la región es pequeña, N puede ser sobre-estimada
y de las especies - Datos de insectos no sirven para predecir aves - Predicción a especies que sean detectados de manera similar con la metodología usada.
.footnote[Guillera-Arroita, G, Kéry, M, Lahoz-Monfort, JJ. Inferring species richness using multispecies occupancy modeling: Estimation performance and interpretation. Ecol Evol. 2019; 9: 780– 792. https://doi.org/10.1002/ece3.4821] ]
4. Estimador clásico
¿Será mejor que un estimador no-paramétrico?
# Riqueza con Chao2----
# Formatear los datos a un vector de frecuencia
inci_Chao <- ylist %>% # historias de captura
map(~rowSums(.,na.rm = T)) %>% # sumo las detecciones en cada sitio
reduce(cbind) %>% # unimos las listas
t() %>% # trasponer la tabla
as_tibble() %>% #formato tibble
mutate_if(is.numeric,~(.>=1)*1) %>% #como es incidencia, formateo a 1 y 0
rowSums() %>% # ahora si la suma de las incidencias en cada sitio
as_tibble() %>%
add_row(value= 67, .before = 1) %>% # el formato requiere que el primer valor sea el número de sitios
as.matrix() # Requiere formato de matriz
# Calcular la riqueza con estimadores no paramétricos
chao_sp <- ChaoSpecies(inci_Chao, datatype = "incidence_freq")
NIChao <- chao_sp$Species_table[4,c(1,3,4)] # Extraer valores de IChao
Nocu<- mcmc_intervals(fit.commu,
pars = "Ntotal",
prob = 0.95,
prob_outer = 0.99,
point_est = "mean")[[1]] %>% # Extraer valores del bayes plot
select(m,l,h) %>% # Seleccionar columnas
rename("Estimate"= m, # Renombrarlas
"95%Lower"= l,
"95%Upper"= h)
Veamos de manera gráfica que tanto difieren las estimaciones de la riqueza
# Unir en un solo dataframe
Nplotdata <- rbind(IChao=NIChao, DR.mod=Nocu) %>%
as.data.frame() %>%
rownames_to_column(.)
# Gráfico para comparar la riqueza estimada
plotN <- ggplot(Nplotdata, aes(x=rowname, y= Estimate, col=rowname))+
geom_point(aes(shape=rowname),size=3)+
geom_errorbar(aes(ymin= `95%Lower`, ymax= `95%Upper`),
width=.3, size=1)+
labs(x="Estimador de riqueza",
y="Número de especies estimado",
title = "Diferencia de los estimadores de riqueza")+
theme_classic()+
theme(text=element_text(size = 13),
plot.title = element_text(hjust= 0.5),
legend.position = "none")
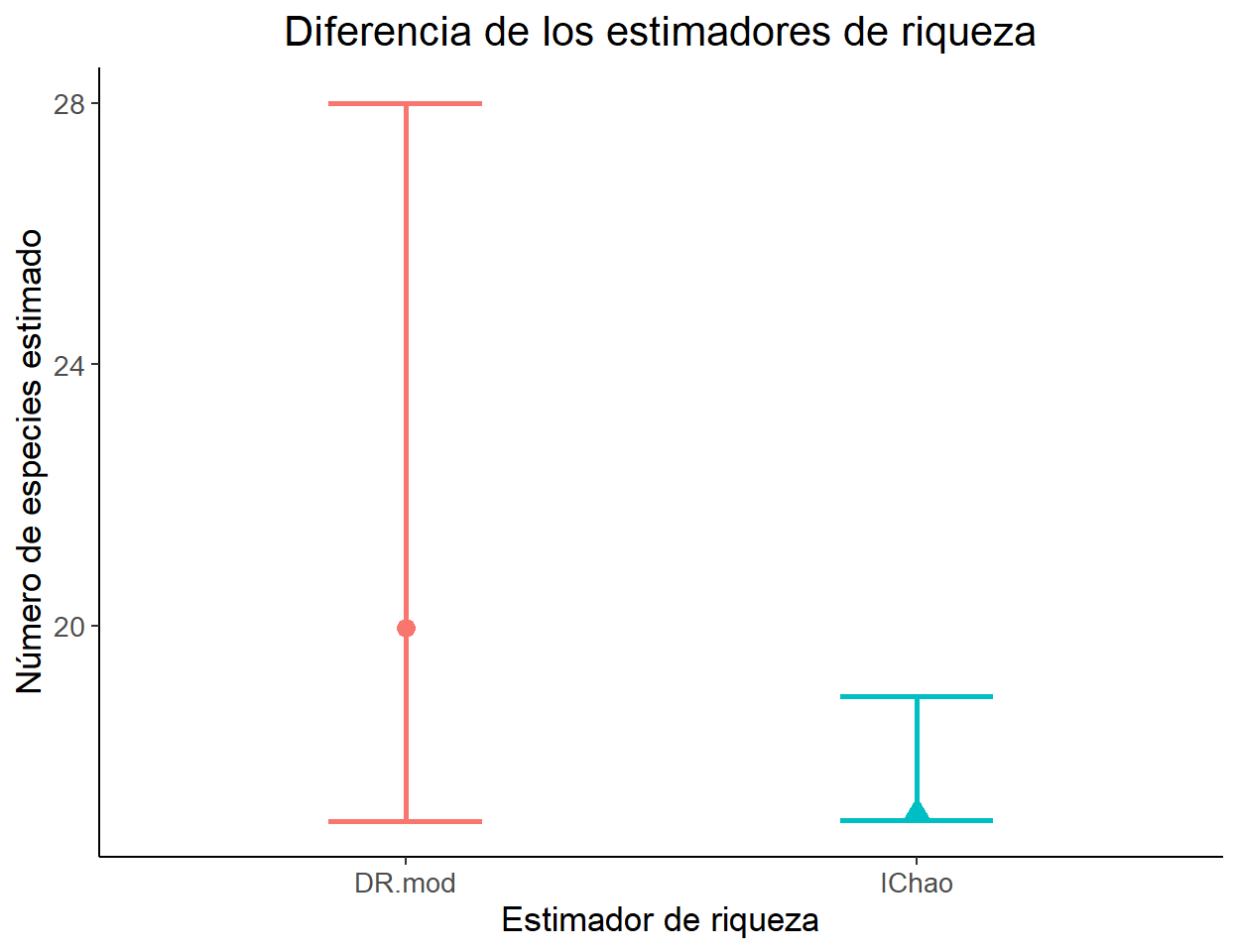
Ocupación multi-specie vs IChao

.footnote[ Tingley, MW, Nadeau, CP, Sandor, ME. Multi-species occupancy models as robust estimators of community richness. Methods Ecol Evol. 2020; 11: 633– 642. https://doi.org/10.1111/2041-210X.13378]
Ambos lo hacen muy mal cuando \(\psi_k\) es muy bajo
- Chao siempre estará sesgado al valor más bajo de N por no considerar la detección (De hecho Chao lo reconoce)
- Los modelos de ocupación son muy hambrientos de datos
- Tienes que saber “programar” para ajustar modelos multi-specie
- Todavía hay algunos bmoles con la heterogeneidad y los prior (Guillera-Arroita et al. 2019)
5.1 Otros ejemplos
Las posibilidades son infinitas 1. Estructura de la diversidad- Número efectivo de especies 
- Estructura de la diversidad- Número efectivo de especies
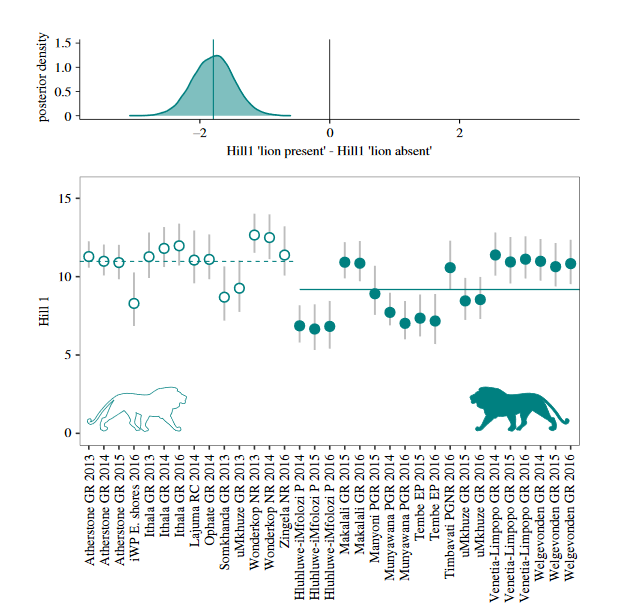
Cómo la presencia o no de leones afecta la riqueza de meso-carnívoros
Curveira-Santos Gonçalo, Sutherland Chris, Tenan Simone, Fernández-Chacón lbert, Mann Gareth K. H., Pitman Ross T.and Swanepoel Lourens H. 2021. Mesocarnivore community structuring in the presence of Africa’s apex predator. Proc. R. Soc. B.2882020237920202379. http://doi.org/10.1098/rspb.2020.2379
- Diversidad funcional y filogenética
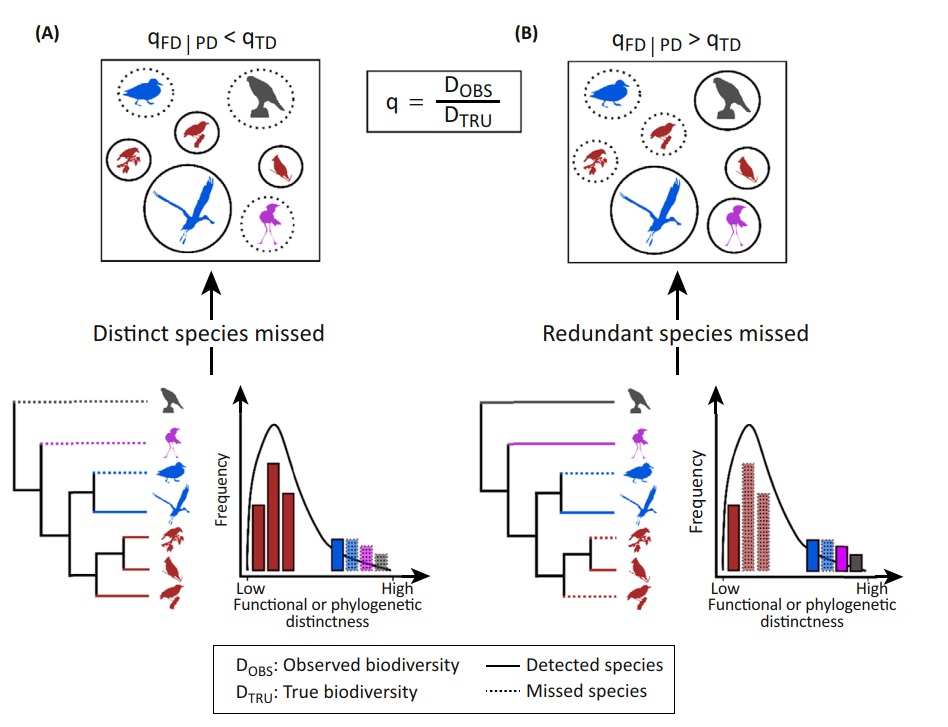
Las consecuencias de no considerar a especies no detectadas en los análisis.
Jarzyna, M. A., & Jetz, W. (2016). Detecting the multiple facets of biodiversity. Trends in ecology & evolution, 31(7), 527-538. https://doi.org/10.1016/j.tree.2016.04.002
Les dejo ejemplos divertidos en la carpeta de bibliografía