Script DivOC_script.R
1. Instalar y cargar el paquete —-
Comenzaremos usando la paquetería DiversityOccupancy. Este paquete estima la diversidad alfa por medio de modelos jerárquicos.
# install.packages("DiversityOccupancy")
library(DiversityOccupancy)
library(camtrapR) #Funciones de registro de especies
library(tidyverse) # Manípular datos y gráficos
library(hillR) # Estimar diversidad
library(ggeffects) #gráficas de pred para glm
library(beepr) # Opcional para avisar R termine
library(tictoc) # Opcional para tomar el tiempo de la función
Este paquete te va a pedir instalar también MuMIn, unmarked, reshape, lattice, Rcpp
2. Cargar los datos —-
Formato de los datos
Son necesarios eventos de muestreo repetidos. Vamos a trabajar con una base de datos de 17 especies, 67 sitios (cámaras) y 19 eventos de muestreo.
Nota: Todas las especies deben tener una matriz de historias de detección de las mismas dimensiones
# Cargamos la tabla de registros de las especies
registers <- read.csv("Data/Survey/recordTable_OC.csv")
table(registers$Species)
Bassariscus astutus Canis latrans
3 6
Canis lupus familiaris Capra hircus
4 13
Conepatus leuconotus Dasypus novemcinctus
79 1
Lynx rufus Mephitis macroura
50 18
Nasua narica Odocoileus virginianus
4 227
Pecari tajacu Procyon lotor
71 40
Puma yagouaroundi Roedores
6 66
Spilogale angustigrons Sylvilagus floridanus
13 492
Urocyon cinereoargenteus
173 # Cargamos la tabla de operación de cámaras
CToperation <- read.csv("Data/Survey/CTtable_OC.csv")
# Generamos la matríz de operación de las cámaras
camop <- camtrapR::cameraOperation(CTtable= CToperation, # Tabla de operación
stationCol= "Station", # Columna que define la estación
setupCol= "Setup_date", #Columna fecha de colocación
retrievalCol= "Retrieval_date", #Columna fecha de retiro
hasProblems= T, # Hubo fallos de cámaras
dateFormat= "%Y-%m-%d") # Formato de las fechas
# Función para generar las historias de detección para todas las especies seleccionadas
DetHist_list <- lapply(unique(registers$Species), FUN = function(x) {
detectionHistory(
recordTable = registers, # Tabla de registros
camOp = camop, # Matriz de operación de cámaras
stationCol = "Station",
speciesCol = "Species",
recordDateTimeCol = "DateTimeOriginal",
recordDateTimeFormat = "%d/%m/%Y",
species = x, # la función reemplaza x por cada una de las especies
occasionLength = 10, # Colapso de las historias a 10 días
day1 = "station", #inicie en la fecha de cada estación
datesAsOccasionNames = FALSE,
includeEffort = TRUE,
scaleEffort = TRUE,
timeZone = "America/Mexico_City"
)}
)
# Se genera una lista con cada historia de detección y el esfuerzo de muestreo, ahora le colocaremos los nombres para saber a cual especie corresponde
names(DetHist_list) <- unique(registers$Species)
# Finalmente creamos una lista nueva donde estén solo las historias de detección
ylist <- lapply(DetHist_list, FUN = function(x) x$detection_history)
# Todas las historias deben tener el mismo número de sitios y de ocasiones de muestreo
data <- ylist %>% # Lista con los datos
reduce(cbind) # Unimos las historias de captura en un solo dataframe
Obtenemos una matriz con el mismo número de sitios 67 y 19 eventos X 17 especies = 323 columnas
| o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | o1 | o2 | o3 | o4 | o5 | o6 | o7 | o8 | o9 | o10 | o11 | o12 | o13 | o14 | o15 | o16 | o17 | o18 | o19 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| C1T2P1 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 1 | 1 | 1 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 1 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| C1T2P11 | NA | NA | NA | 1 | 0 | 1 | 0 | 1 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 1 | 0 | 1 | 1 | 1 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 1 | 1 | 0 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 1 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 1 | 0 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| C1T2P21 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 1 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 1 | 1 | 0 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 1 | 1 | 1 | 0 | 1 | 0 | 0 | 1 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| C1T4P1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 1 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| C1T4P11 | 1 | 0 | 1 | 0 | 0 | 0 | 1 | 1 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 1 | 1 | 1 | 1 | 1 | 0 | 1 | 1 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 1 | 1 | 1 | 1 | 0 | 1 | 0 | 1 | 1 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 1 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 1 | 0 | 1 | 1 | 0 | 1 | 1 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA |
| C1T4P21 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | NA | NA | NA | NA | NA | NA | NA | NA | NA |
Cargar las covariables
Vamos a leer el archivo .csv de la ruta data/Covs/ que contiene las covariables de sitio (no usaremos de observación). Todas estas estandarizadas
| Vertcover_50 | Dcrops | MSAVI | Slope | Effort | Dpop_G |
|---|---|---|---|---|---|
| -0.4744602 | 0.6765368 | 0.7316819 | -0.2195550 | -0.2080680 | -0.4260974 |
| 0.6519157 | 1.4620448 | -0.8666152 | -0.9927301 | -1.4605398 | -0.3167450 |
| -0.9243340 | 2.2193059 | -0.5838870 | -1.0258694 | -0.0719298 | -0.1805434 |
| -0.9243340 | 0.4603495 | -0.1430110 | 1.4183658 | -0.8887592 | -0.1922714 |
Importante: Cada proceso es afectado por diferentes covariables. Para más información mira este enlace
3. Modelos de abundancia —-
3.1 Modelos básicos —-
Vamos a utilizar la función diversityoccu(). Se genera un objeto lista con los modelos para cada una de las especies, cálculo de diversidad y otras cosas….
cam_diver <- diversityoccu(pres = data, # La matriz de datos
sitecov = covars, # Covariables de sitio
obscov = NULL, # no tenemos covariables de observación,
spp = 17, # Número de especies
form = ~ Effort + Slope ~ Dcrops, # Formula del modelo p(), lambda()
dredge = FALSE # En este primer ejemplo no usaremos AIC
)
[1] "Species 1 ready!"
[1] "Species 2 ready!"
[1] "Species 3 ready!"
[1] "Species 4 ready!"
[1] "Species 5 ready!"
[1] "Species 6 ready!"
[1] "Species 7 ready!"
[1] "Species 8 ready!"
[1] "Species 9 ready!"
[1] "Species 10 ready!"
[1] "Species 11 ready!"
[1] "Species 12 ready!"
[1] "Species 13 ready!"
[1] "Species 14 ready!"
[1] "Species 15 ready!"
[1] "Species 16 ready!"
[1] "Species 17 ready!"Se va a generar un objeto lista con los modelos para cada una de las especies, cálculo de diversidad y otras cosas….
Veamos uno de los modelos 🐺
cam_diver$models[[9]] # Modelo para la especie 9
Call:
occuRN(formula = form, data = data2)
Abundance:
Estimate SE z P(>|z|)
(Intercept) -0.1543 0.278 -0.555 0.579
Dcrops -0.0306 0.190 -0.161 0.872
Detection:
Estimate SE z P(>|z|)
(Intercept) -2.835 0.339 -8.37 5.54e-17
Effort 0.909 0.324 2.81 4.99e-03
Slope -0.596 0.216 -2.76 5.78e-03
AIC: 317.6726 No sabemos si todas las especies responden de la misma manera a las covariables que usamos. Debemos escoger de todas las variables cual se ajusta mejor a cada especie
¿Cómo vamos a generar todas las posibles combinaciones de modelos para cada especie?
3.2 Mejores modelos con AIC —-
Tranquilo esta misma función lo hace por ti :D. Solamente tenemos que activar dredge
Dependiendo de tu computador la función puede tardar más o menos. En la mía duró ~57 segs. Pero puede tardar mucho más dependiendo del número de especies, sitios, eventos de muestreo y cantidad de covariables.
cam_diver_AIC <- diversityoccu(pres = data, # La matriz de datos
sitecov = covars, # Covariables de sitio
obscov = NULL, # no tenemos covariables de observación,
spp = 17, # Número de especies
form = ~ Effort + Slope ~ Dcrops, # Formula del modelo p(), lambda()
dredge = TRUE # escoge los mejores modelos con AIC
)
Veamos de nuevo el modelo que seleccionó para la sp9 🐺
cam_diver$models[[9]]
Call:
occuRN(formula = form, data = data2)
Abundance:
Estimate SE z P(>|z|)
(Intercept) -0.1543 0.278 -0.555 0.579
Dcrops -0.0306 0.190 -0.161 0.872
Detection:
Estimate SE z P(>|z|)
(Intercept) -2.835 0.339 -8.37 5.54e-17
Effort 0.909 0.324 2.81 4.99e-03
Slope -0.596 0.216 -2.76 5.78e-03
AIC: 317.6726 cam_diver_AIC$models[[9]]
Call:
occuRN(formula = ~Effort + Slope + 1 ~ 1, data = data2)
Abundance:
Estimate SE z P(>|z|)
-0.145 0.272 -0.532 0.595
Detection:
Estimate SE z P(>|z|)
(Intercept) -2.846 0.333 -8.54 1.30e-17
Effort 0.925 0.308 3.01 2.62e-03
Slope -0.596 0.215 -2.77 5.58e-03
AIC: 315.6987 3.3 Gráfico de predicción —-
Veamos el resultado gráfico para la especie 3
(responseplot.abund( batch = cam_diver_AIC, # objeto creado con diversityoccu
spp = 3, # número o nombre de la sp
variable= Dcrops # variable
))
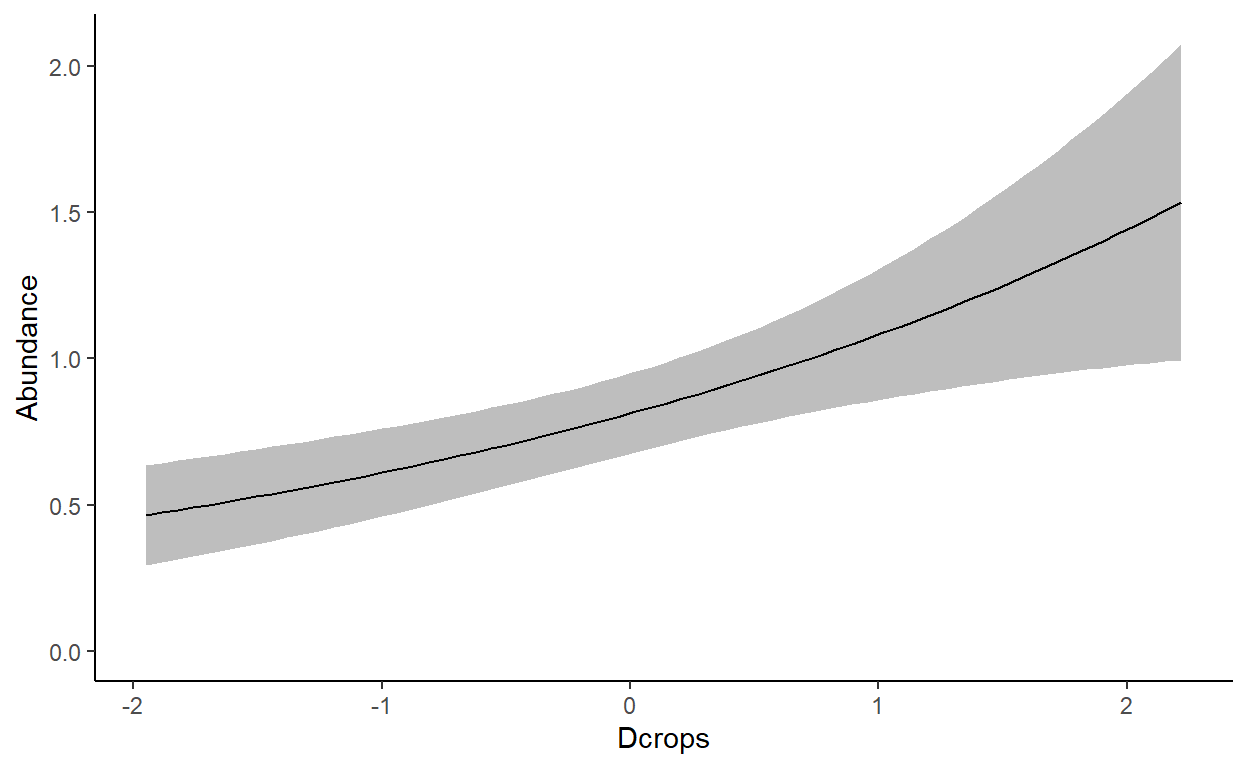
4. Modelando la diversidad —-
Tenemos un modelo donde se estima la abundancia para cada especie, es hora de modelar la diversidad
glm.div <- model.diversity(DivOcc = cam_diver_AIC,# modelos
method = "h", # método
delta = 2,
squared = T # términos cuadráticos
)
AICtab <- glm.div$Table
| model | aicc | weights | Delta.AICc |
|---|---|---|---|
| Diversity ~ 1 + Dcrops + I(Dcrops^2) | -331.8899 | 0.3229081 | 0.0000000 |
| Diversity ~ 1 + Vertcover_50 + Dcrops + I(Dcrops^2) | -331.3285 | 0.2438852 | 0.5613404 |
| Diversity ~ 1 + Dcrops + MSAVI + I(Dcrops^2) | -330.5617 | 0.1662141 | 1.3281821 |
| Diversity ~ 1 + Dcrops + MSAVI + I(Dcrops^2) + I(MSAVI^2) | -330.3166 | 0.1470432 | 1.5732824 |
| Diversity ~ 1 + Dcrops + Dpop_G + I(Dcrops^2) | -329.9093 | 0.1199495 | 1.9805941 |
4.1 Respuesta Gráfica de la diversidad a distintas variables —-
Ahora veamos la respuesta gráfica de la diversidad a una variable
responseplot.diver(glm.div, Dcrops)
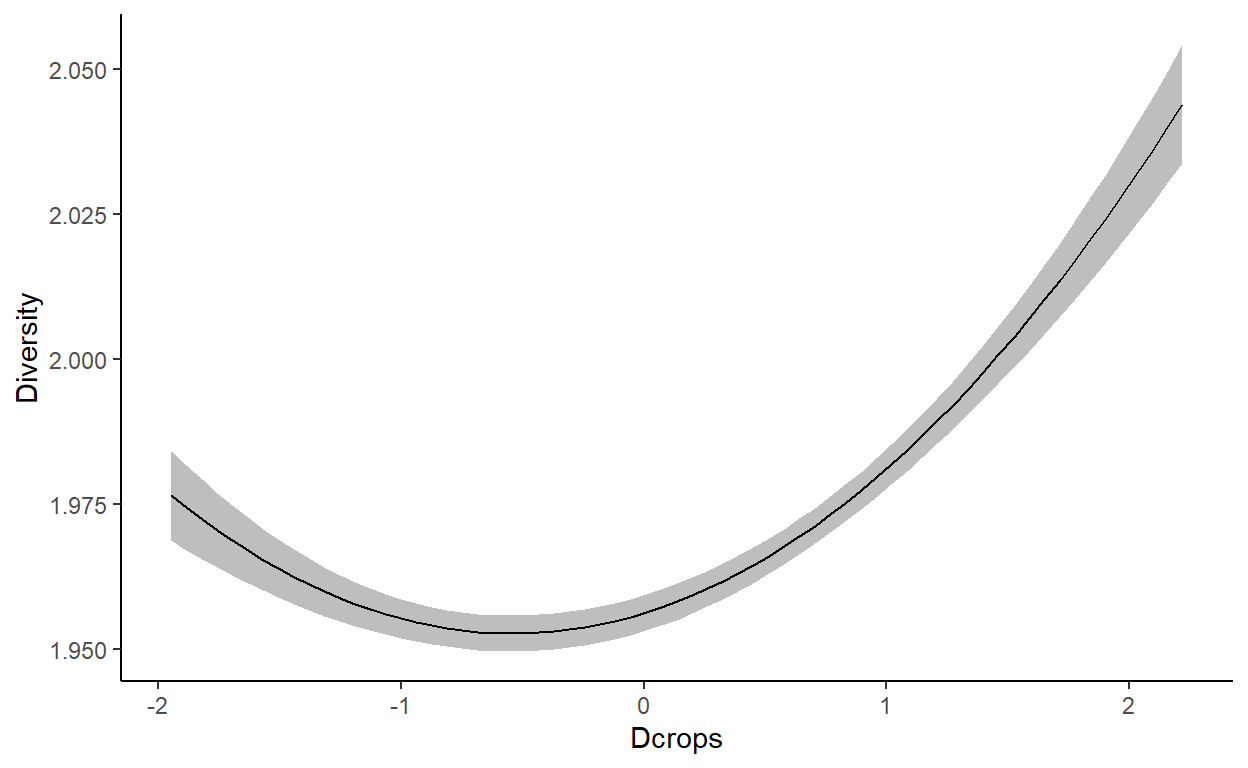
A medida que aumenta el valor de distancia a cultivos (escalado) hay mayor diversidad ?????
Relativamente fácil, para ser verdad …..

Si seguimos la viñeta del paquete nunca nos dice que elemento de la diversidad mide o calcula..
- Riqueza?
- Shannon, simpson?….
- Número efectivo de especies?? (Hill)
Glm? de que tipo?
Un Glm puede ser de varias familias (distribuciones) y depende de la naturaleza de los datos: conteos, proporciones, unos y ceros
Es importante leer el manual
4.2 Diversidad para cada indice de entropía
Hay otro argumento de la función diversityoccu y es “index”. Este argumento permite escoger que índice utilizar “shannon”, “simpson” o “invsimpson”.
cam_diver_sh <- diversityoccu(pres = data,
sitecov = covars,
obscov = NULL,
spp = 17,
form = ~ Effort + Slope ~ Dcrops,
dredge = TRUE,
index = "shannon" #<<
)
Podemos aplicar la función para cada índice…?
También se puede hacer una función que lo haga en automático, pero por simplicidad (
no se hacerlo bien) corremos tres veces la función
cam_diver_sim <- diversityoccu(pres = data,
sitecov = covars,
obscov = NULL,
spp = 17,
form = ~ Effort + Slope ~ Dcrops, dredge = TRUE,
index = "simpson" #<<
)
cam_diver_inv <- diversityoccu(pres = data,
sitecov = covars,
obscov = NULL,
spp = 17,
form = ~ Effort + Slope ~ Dcrops, dredge = TRUE,
index = "invsimpson" #<<
)
Índices de entropía …..
El problema de estos índices (para los ecólogos) es que - son adimensionales - no siguen una relación lineal con la riqueza (doble de riqueza \(\neq\) doble de diversidad)
5. Calculamos número efectivo de especies —-
Calculemos el número efectivo de especies con las abundancias estimadas
Sospechoso…
| species.1 | species.2 | species.3 | species.4 | species.5 | species.6 | species.7 | species.8 | species.9 | species.10 | species.11 | species.12 | species.13 | species.14 | species.15 | species.16 | species.17 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0.6082829 | 1.179013 | 0.9865411 | 0.7986634 | 0.6344764 | 0.470707 | 1.73449 | 2.942110 | 0.8654214 | 0.1201388 | 8.197757 | 0.1485776 | 8.906907 | 0.0098931 | 0.119418 | 11.42479 | 0.237951 |
| 0.6082829 | 1.179013 | 1.2351996 | 0.7986634 | 0.6344764 | 0.470707 | 1.73449 | 6.441039 | 0.8654214 | 0.1201388 | 8.197757 | 0.1485776 | 8.906907 | 0.0014365 | 0.119418 | 11.42479 | 0.237951 |
| 0.6082829 | 1.179013 | 1.5340820 | 0.7986634 | 0.6344764 | 0.470707 | 1.73449 | 13.709316 | 0.8654214 | 0.1201388 | 8.197757 | 0.1485776 | 8.906907 | 0.0002236 | 0.119418 | 11.42479 | 0.237951 |
| 0.6082829 | 1.179013 | 0.9273586 | 0.7986634 | 0.6344764 | 0.470707 | 1.73449 | 2.371387 | 0.8654214 | 0.1201388 | 8.197757 | 0.1485776 | 8.906907 | 0.0168258 | 0.119418 | 11.42479 | 0.237951 |
| 0.6082829 | 1.179013 | 1.1369200 | 0.7986634 | 0.6344764 | 0.470707 | 1.73449 | 4.824359 | 0.8654214 | 0.1201388 | 8.197757 | 0.1485776 | 8.906907 | 0.0029269 | 0.119418 | 11.42479 | 0.237951 |
| 0.6082829 | 1.179013 | 1.4022314 | 0.7986634 | 0.6344764 | 0.470707 | 1.73449 | 10.022276 | 0.8654214 | 0.1201388 | 8.197757 | 0.1485776 | 8.906907 | 0.0004836 | 0.119418 | 11.42479 | 0.237951 |
Calculemos diversidad con hillR
Ahora unimos las bases de datos y las covariables para modelar
# Unir las bases de perfiles de diversidad
hill_div <- data.frame(q0=q0, q1=q1, q2=q2)
# Unir con las covariables
glm_hill <- cbind(hill_div, covars)
Obtenemos esta base
| q0 | q1 | q2 | Vertcover_50 | Dcrops | MSAVI | Slope | Effort | Dpop_G |
|---|---|---|---|---|---|---|---|---|
| 17 | 7.290641 | 5.283733 | -0.4744602 | 0.6765368 | 0.7316819 | -0.2195550 | -0.2080680 | -0.4260974 |
| 17 | 7.476420 | 5.687832 | 0.6519157 | 1.4620448 | -0.8666152 | -0.9927301 | -1.4605398 | -0.3167450 |
| 17 | 7.068294 | 5.418051 | -0.9243340 | 2.2193059 | -0.5838870 | -1.0258694 | -0.0719298 | -0.1805434 |
| 17 | 7.204272 | 5.173335 | -0.9243340 | 0.4603495 | -0.1430110 | 1.4183658 | -0.8887592 | -0.1922714 |
| 17 | 7.448817 | 5.558398 | -1.1509622 | 1.1723154 | 0.2129457 | -0.0597006 | -0.0719298 | -0.0948151 |
| 17 | 7.342298 | 5.686248 | 0.6519157 | 1.9052628 | 0.5284530 | -1.0258694 | -0.0719298 | 0.0268169 |
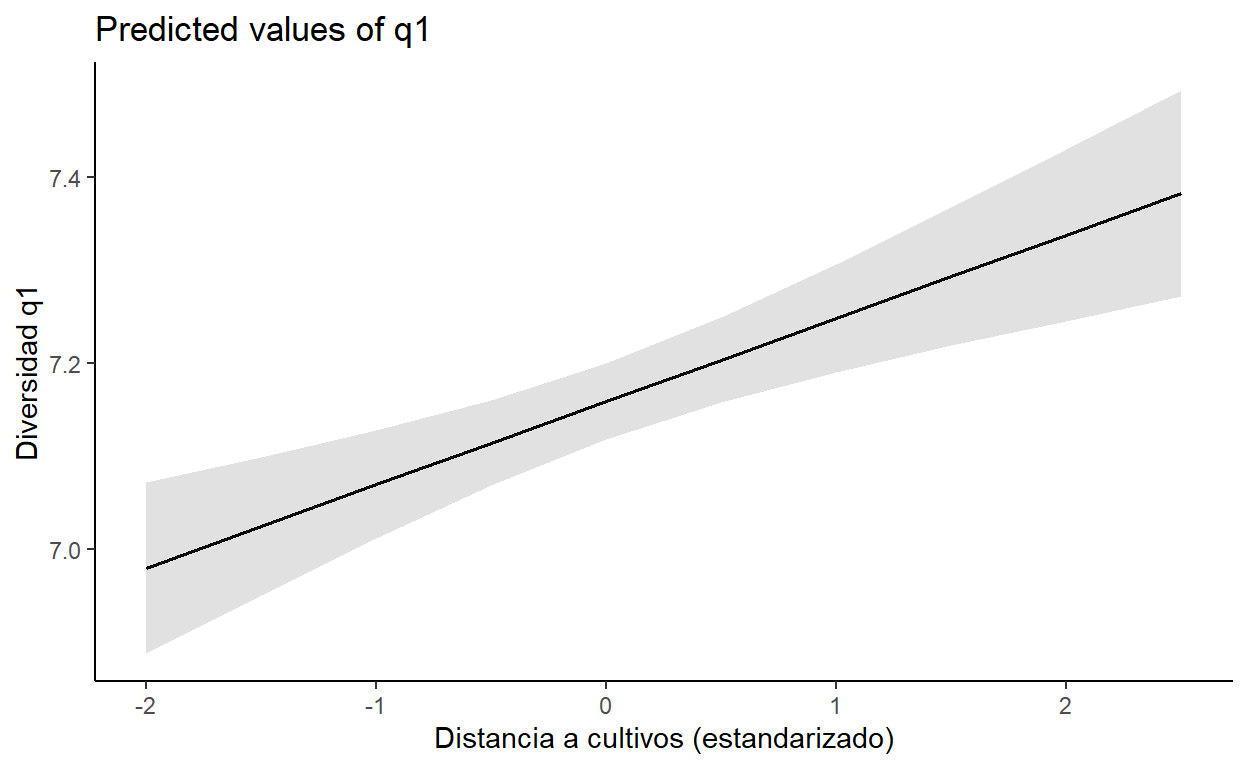
Ahora ajustemos un glm sencillo
glm_q1 <- glm(q1~ Dcrops, family = gaussian, data = glm_hill)
y usemos ggeffects para graficar
plot_q1 <- ggpredict(glm_q1, terms = "Dpop_G")
plot(plot_q1)+
labs(y= "Diversidad q1",
x= "Distancia a poblados (estandarizado)")+
theme_classic()
Tarea
- Modelar los otros perfiles de diversidad
- Crear modelos candidatos con otras covariables y escoger con AIC

6. Consideraciones finales
El paquete es bueno y agiliza muchos pasos del modelado
Cuando no hay ninguna covariable que explique la “abundancia”, se asume que es constante para todos los sitios. Eso puede subestimar o sobrestimar la abundancia…
“Abundancia” constante: diversidad de la localidad y no de la cámara?
Si se usan los índices de entropía se debe saber cómo interpretarlos (Jost 2006 ; Jost et al 2010)]
7. Ejemplo en una investigación
Un ejemplo de cómo se pueden usar los modelos Royle-Nichols para calcular diversidad
Tesis de licenciatura pregrado premiada por la Asosiación Mexicana de Mastozoología A.C. (AMMAC)
https://www.youtube.com/watch?v=qaD9NRAg3SQ ]
FIN