library(ggplot2) # Carguemos ggplo2
#install.packages("palmerpenguins")
library(palmerpenguins) # Cargar el paqueteUna de las cosas más divertidas y potentes de R es su capacidad gráfica. En esta sección vamos a explorar algunos gráficos sencillos de R base y su versión en ggplot2. Cuando me refiero a base R es la función de plot que viene con R. Mientras que ggplot2 es una sintaxis especializada para generar gráficos por capas en R. No hay un camino correcto en este caso, lo mejor es que cada quien use el método que más entienda o que más le llame la atención. Yo personalmente entiendo mejor (y se más trucos) al ggplot.
Datos
Vamos a usar unos datos especiales para realizar la mayoría de las gráficas. Estos datos vienen en un paquete de R llamado palmerpenguins y si, es sobre pingüinos. Para usarlo tenemos que instalarlo y luego cargar los datos
Nubes de puntos
plot(x= penguins$flipper_length_mm, # Definir eje x
y= penguins$body_mass_g, # Definir eje y
pch= 18, # Tipo de punto
cex= 2, # Tamaño
col= "#69b3a2", # Color
xlab = "Largo de aleta (mm)", # Nombre eje x
ylab= "Masa corporal (g)", # Nombre eje y
main= "Tamaño de pingüinos") # Título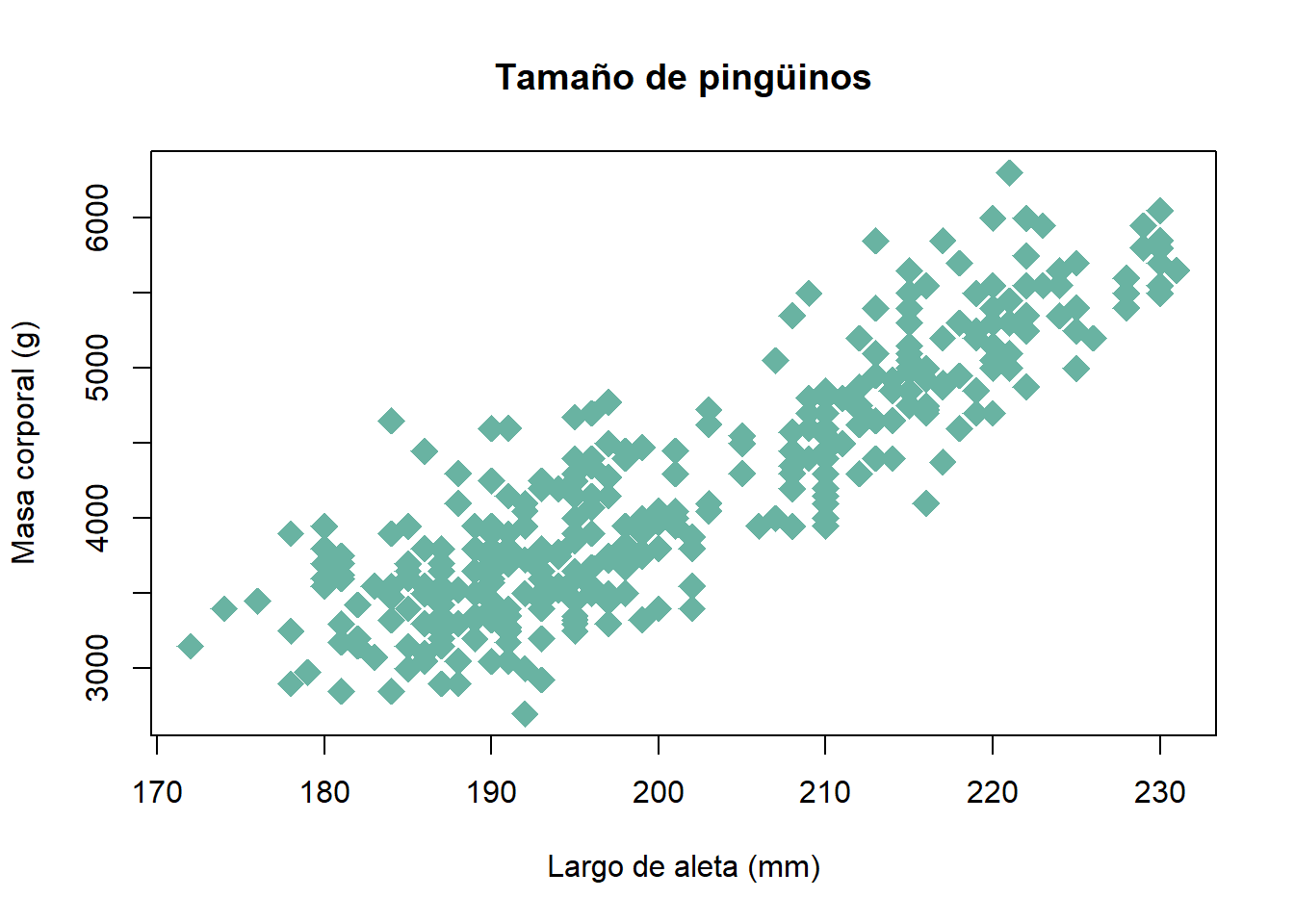
ggplot(data = penguins, # Definir datos
aes(x=flipper_length_mm, # Eje x
y = bill_length_mm))+ # Eje y
geom_point(shape= 18, # Tipo de punto
col= "#69b3a2", # Color
size= 3)+ # Tamaño
labs(title= "Tamaño de pingüinos", #título
x= "Largo de aleta (mm)", # Leyenda eje x
y= "Masa corporal (g)")+ # Leyenda eje y
theme_bw() # Adicional de diseñoWarning: Removed 2 rows containing missing values or values outside the scale range
(`geom_point()`).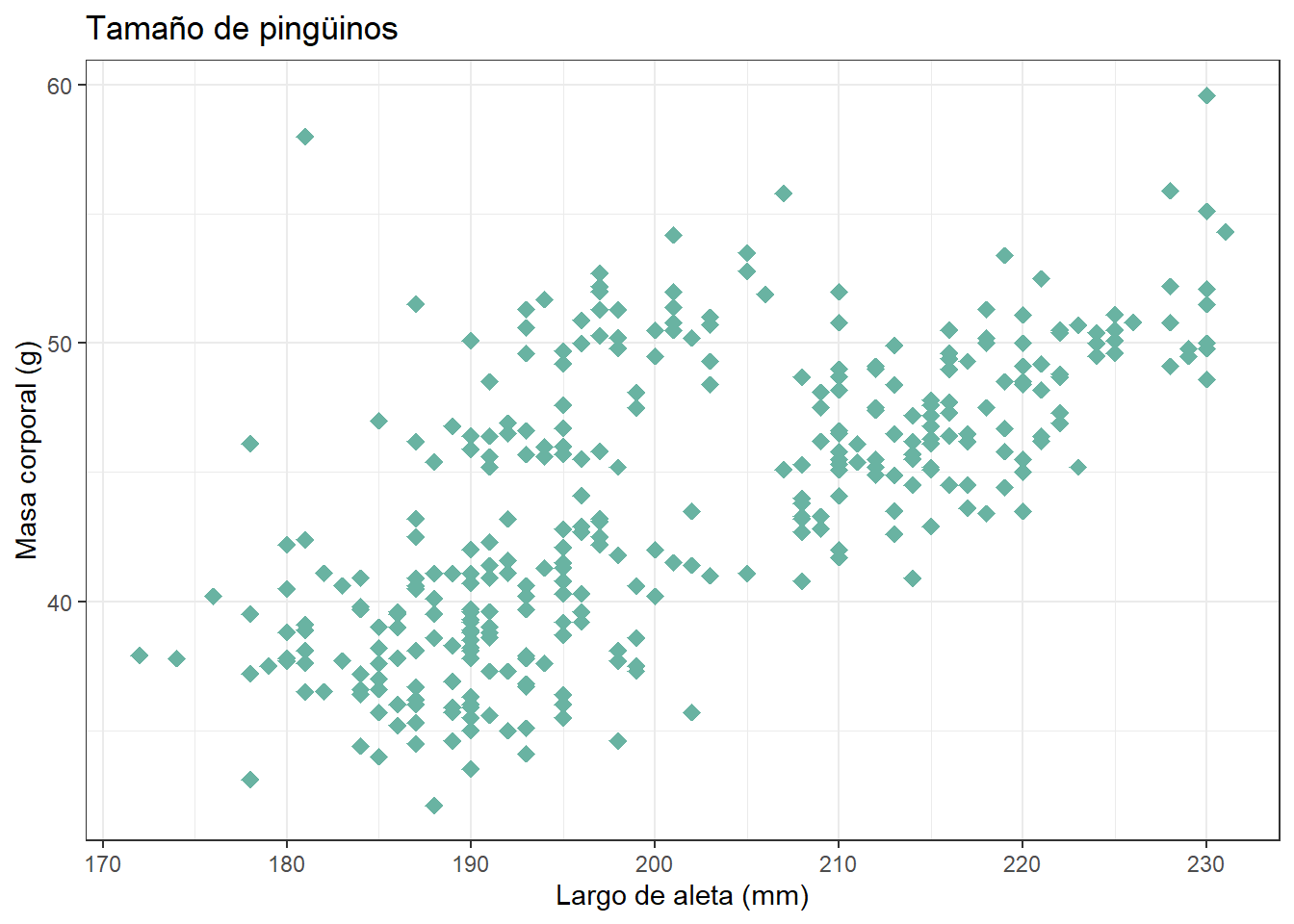
Histogramas
hist(penguins$bill_depth_mm, # Datos
prob= TRUE, # False es de frecuencia
breaks = 10, # Número de segmentos
col= "#E1DEFC", # Color
border = "#9A68A4", # Borde
main= "Histograma con plot",
xlab= "Alto del pico",
ylab= "Densidad")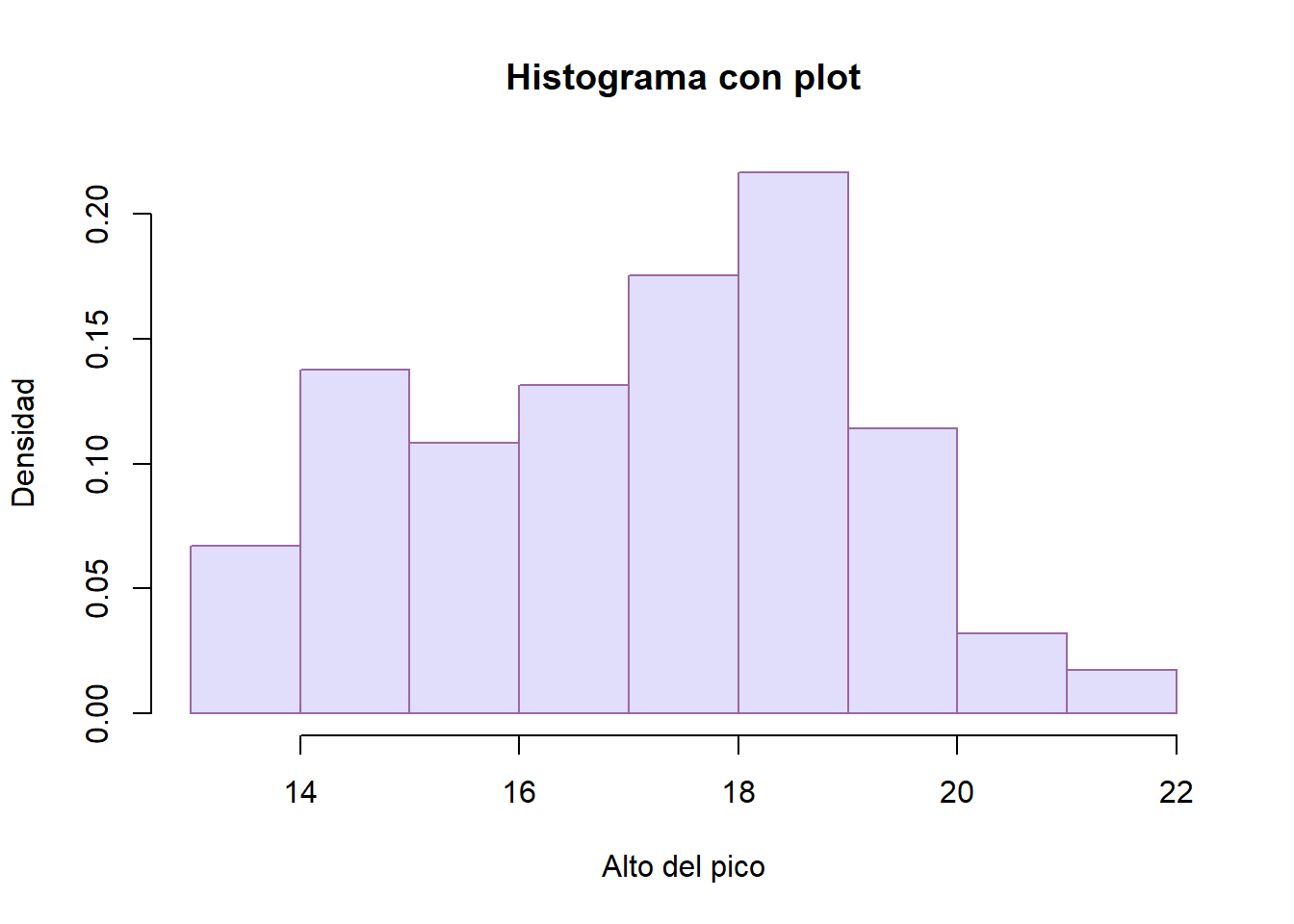
ggplot(data = penguins, # Definir datos
aes(x=bill_depth_mm))+ # Eje x
geom_histogram(aes(y=..density..), #Definir densidad
fill= "#E1DEFC", # Color
col= "#9A68A4", # Borde
bins = 10)+ # Número de segmentos
labs(title= "Histograma con ggplot",
x= "Alto del pico",
y= "Densidad")+
theme_classic()Warning: The dot-dot notation (`..density..`) was deprecated in ggplot2 3.4.0.
ℹ Please use `after_stat(density)` instead.Warning: Removed 2 rows containing non-finite outside the scale range
(`stat_bin()`).
Gráfico de densidad
# Generar densidades por especie
densidades <- tapply(penguins$bill_length_mm, penguins$species, density, na.rm=TRUE)
# Gráfico
plot(densidades$Adelie, # Llamar densidad de una especie
xlim= c(min(penguins$bill_length_mm, # Definir limites
na.rm = TRUE),# Existencia de NAs
max(penguins$bill_length_mm,
na.rm = TRUE)),
main= "Densidad por especie")# Gráfico principal
lines(densidades$Chinstrap, col=2) # Linea por especie
lines(densidades$Gentoo, col=3)
# Legenda
legend("topright", # Posición
legend = levels(as.factor(penguins$species)) , # Definir niveles
lty = 1, # Tipo de linea
col = 1:3) # Color
ggplot(penguins, aes(x= bill_length_mm))+ # Datos
geom_density(aes(fill= species), # Color por especie
alpha= 0.5)+ # Opacidad
labs("Densidad por especie",
x= "Largo del pico",
fill= "Especies")+
theme_bw()Warning: Removed 2 rows containing non-finite outside the scale range
(`stat_density()`).
Boxplot
boxplot(flipper_length_mm ~ species, # Formula de gráfico
data = penguins, # Datos
col= "white", # Color del boxplot
main= "Boxplot",
ylab= "Largo de aleta (mm)",
xlab= "Species")
stripchart(flipper_length_mm ~ species, # Agregar jitter
data = penguins,
method= "jitter", # metodo de puntos
pch= 19, # tipo de puntos
col= 2:4, # Colores
vertical= TRUE,
add= TRUE) # Adicionar al anterior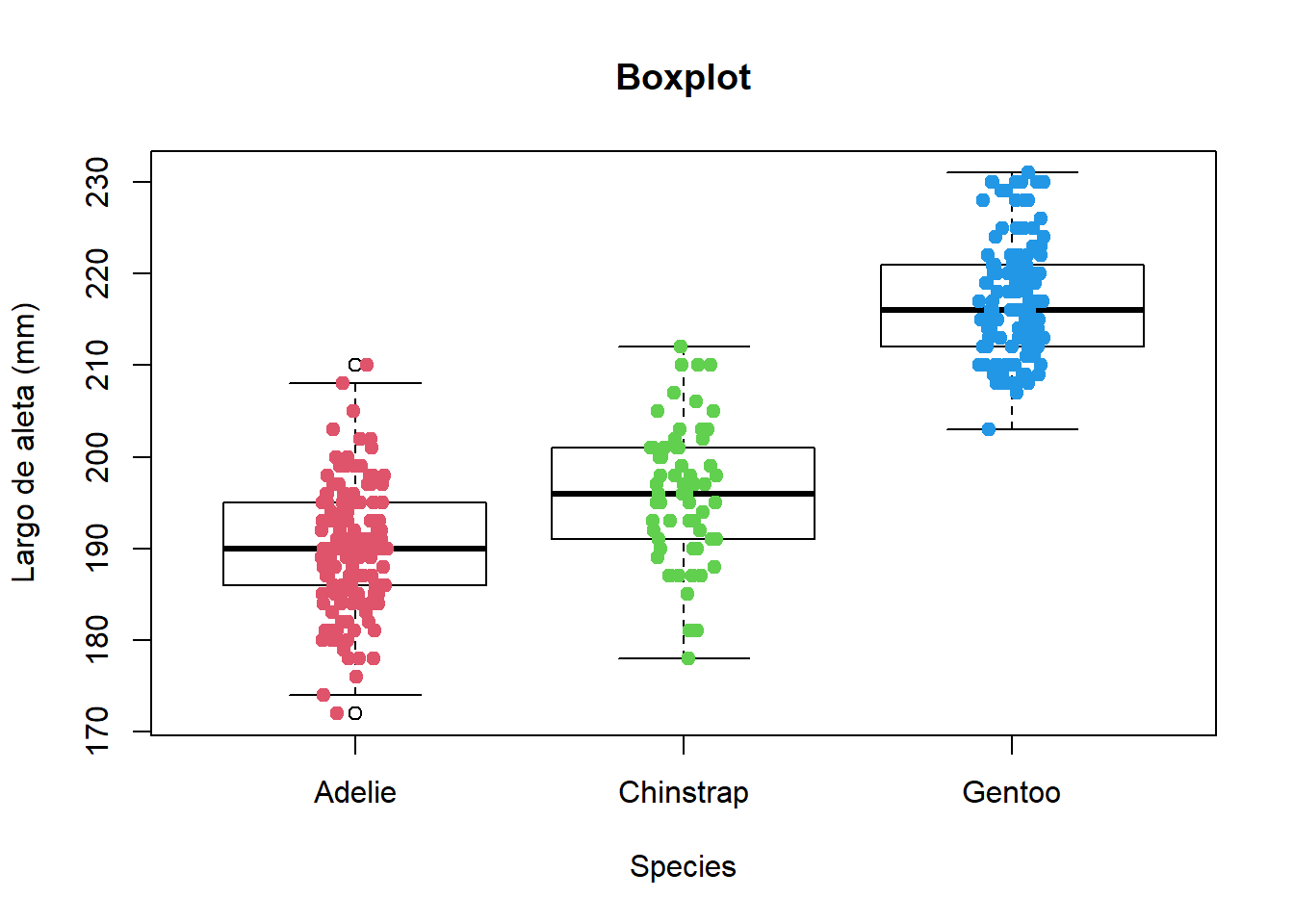
ggplot(penguins, aes(x= species, # Definir x
y= flipper_length_mm))+ #Definit y
geom_boxplot()+ # Agregar boxplot
geom_jitter(aes(col=species), # Agregar puntos y color
width = 0.2)+ #Ancho
labs(title = "Boxplot con ggplot",
y= "Largo de aleta (mm)",
x= "Especies")+
theme_bw()+
theme(legend.position = "none")Warning: Removed 2 rows containing non-finite outside the scale range
(`stat_boxplot()`).Warning: Removed 2 rows containing missing values or values outside the scale range
(`geom_point()`).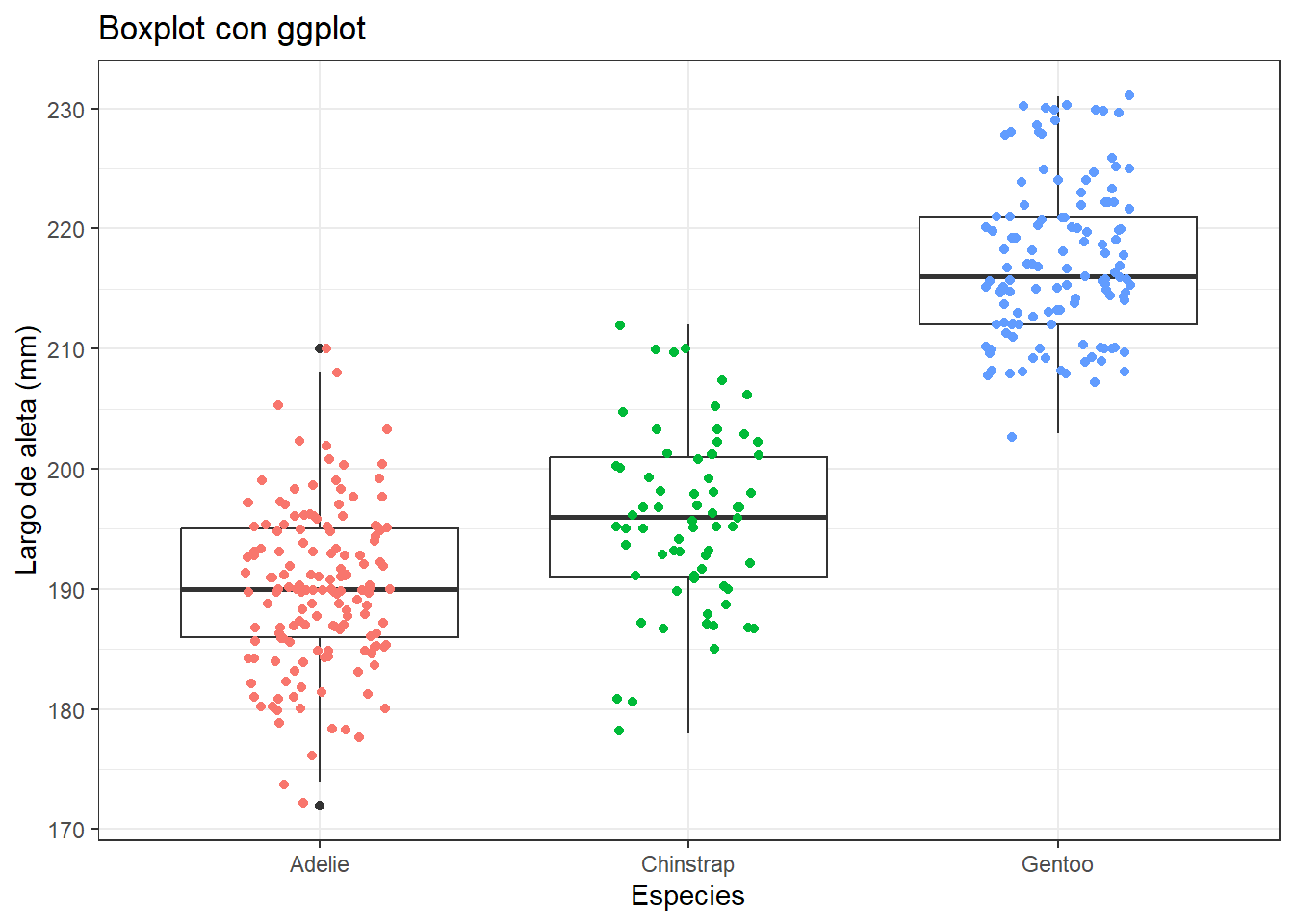
Lineas de regresión
plot(x= penguins$bill_length_mm, #Definir x
y= penguins$bill_depth_mm, #Definit y
pch= 16, # Tipo de punto
xlab= "Alto de pico (mm)",
ylab= "Longitud de pico (mm)")
abline(lm(bill_depth_mm~bill_length_mm, # Agregar linea con base en un lm
data = penguins),
col= 4, # Color de linea
lwd= 3) # Tipo de linea
coef <- round(coef(lm(bill_depth_mm~bill_length_mm,
data = penguins)), # Extraer coeficientes
2)
text(x=36, # coordenadas del texto
y=14, # coordenadas en y del texto
paste("Y = ", coef[1], "+", coef[2], "x")) # Texto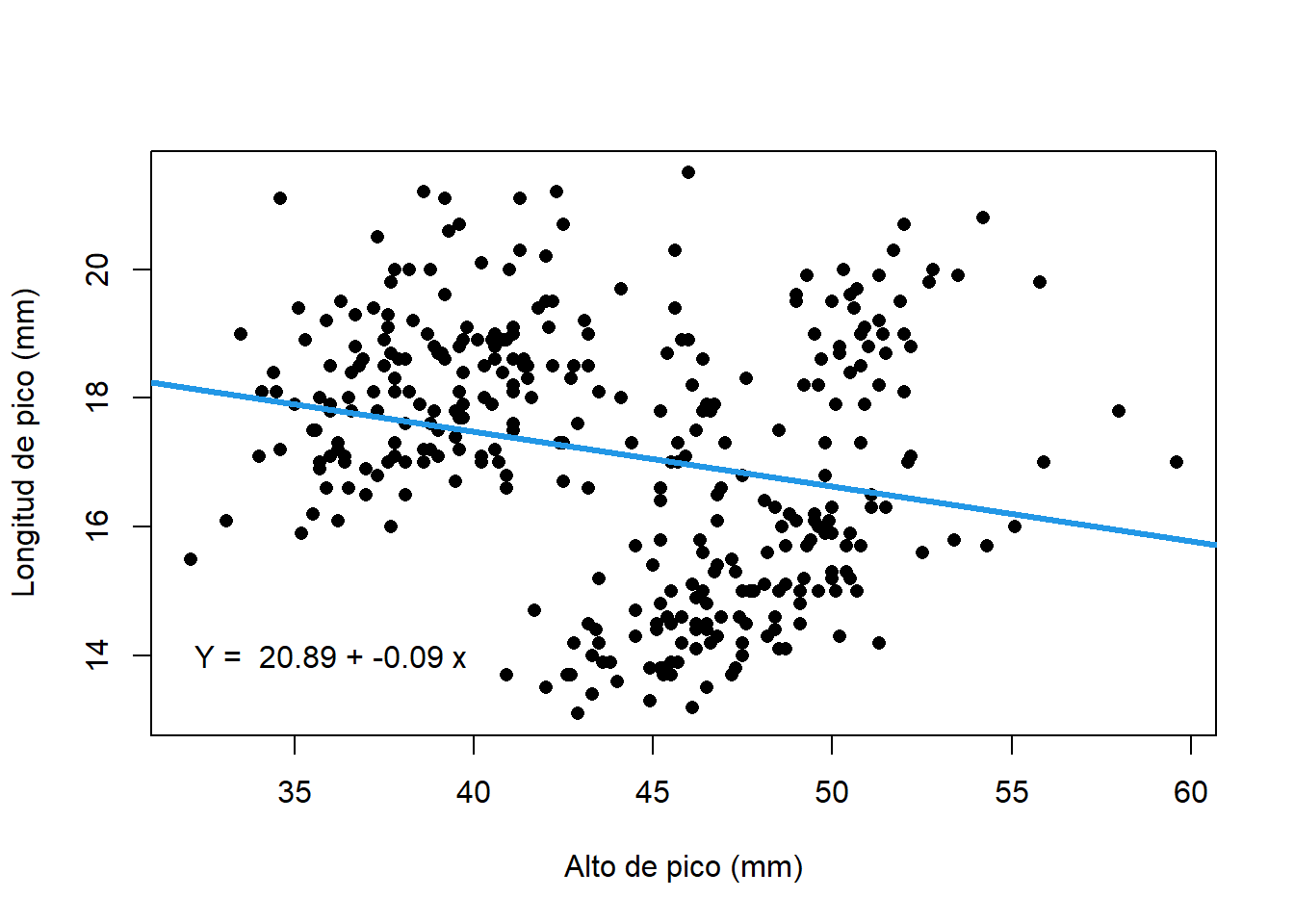
ggplot(penguins, aes(x= bill_length_mm, # Datos en x
y= bill_depth_mm))+ # datos de y
geom_point()+ # Agregar puntos
geom_smooth(method = "lm")+ # Agregar modelo
annotate("text", x=36, y=14, # Texto
label= paste("Y = ", coef[1], "+", coef[2], "x") )+
labs(x= "Alto de pico (mm)",
y= "Longitud de pico (mm)")+
theme_minimal()`geom_smooth()` using formula = 'y ~ x'Warning: Removed 2 rows containing non-finite outside the scale range
(`stat_smooth()`).Warning: Removed 2 rows containing missing values or values outside the scale range
(`geom_point()`).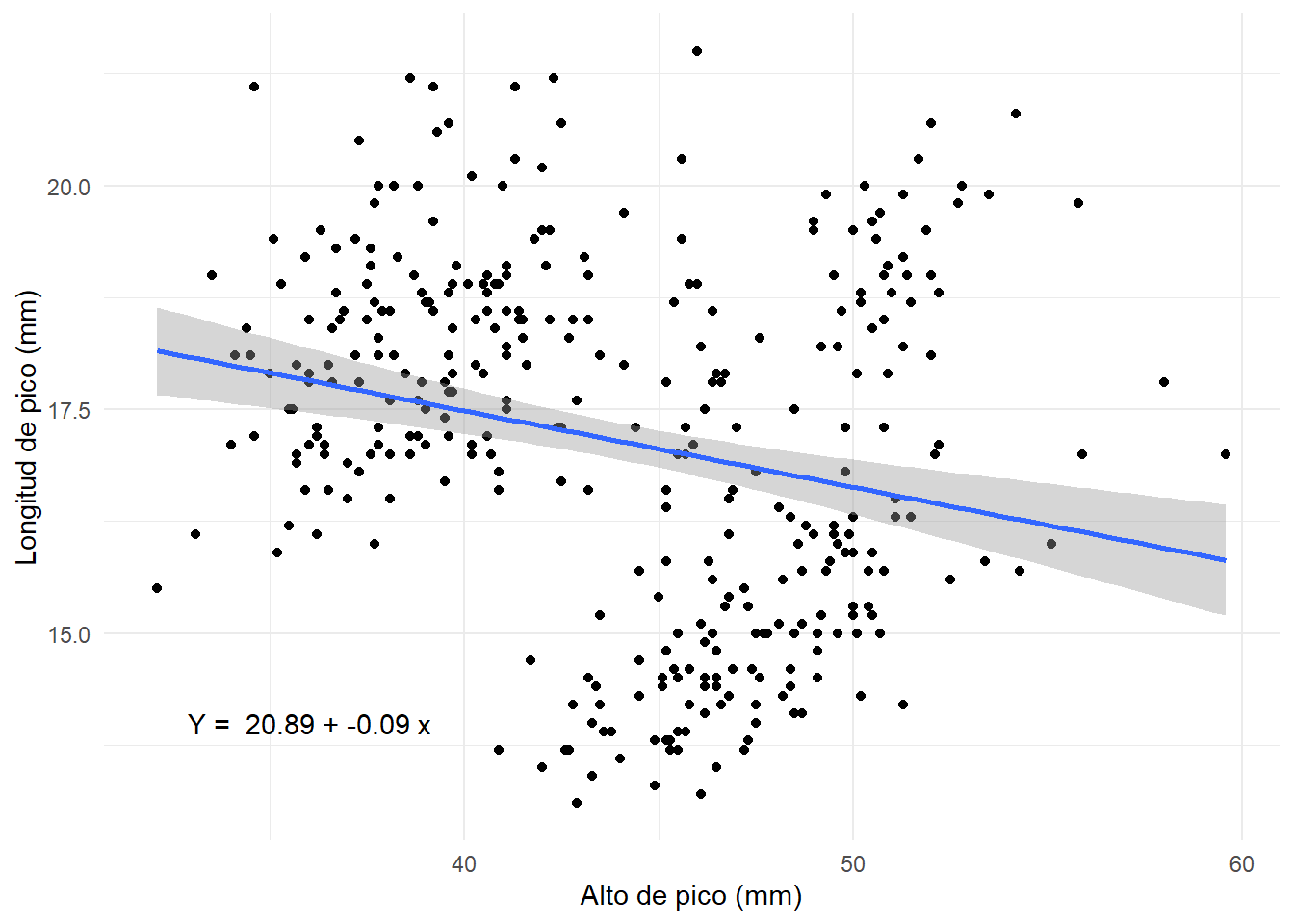
Ejemplo avanzado